Cistrón
(leer más)
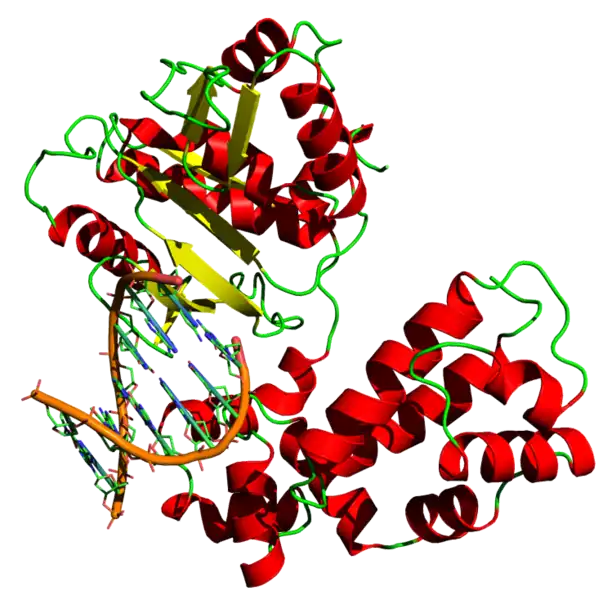
Una ADN polimerasa es miembro de una familia de enzimas que catalizan la síntesis de moléculas de ADN a partir de nucleósidos trifosfatos, los precursores moleculares del ADN. Estas enzimas son esenciales para la replicación del ADN y, por lo general, trabajan en grupos para crear dos dúplex de ADN idénticos a partir de un único dúplex de ADN original. Durante este proceso, la ADN polimerasa "lee" las hebras de ADN existentes para crear dos nuevas hebras que coincidan con las existentes. Estas enzimas catalizan la reacción química.desoxinucleósido trifosfato + ADN n ⇌ pirofosfato + ADN n+1.
La ADN polimerasa agrega nucleótidos a los tres extremos principales (3') de una cadena de ADN, un nucleótido a la vez. Cada vez que una célula se divide, se requieren polimerasas de ADN para duplicar el ADN de la célula, de modo que se pueda pasar una copia de la molécula de ADN original a cada célula hija. De esta manera, la información genética se transmite de generación en generación.
Antes de que pueda tener lugar la replicación, una enzima llamada helicasa desenrolla la molécula de ADN de su forma estrechamente tejida, rompiendo en el proceso los enlaces de hidrógeno entre las bases de nucleótidos. Esto abre o "descomprime" el ADN de doble cadena para dar dos cadenas sencillas de ADN que se pueden usar como moldes para la replicación en la reacción anterior.
En 1956, Arthur Kornberg y sus colegas descubrieron la ADN polimerasa I (Pol I) en Escherichia coli. Describieron el proceso de replicación del ADN mediante el cual la ADN polimerasa copia la secuencia de bases de una hebra de ADN molde. Kornberg recibió más tarde el Premio Nobel de Fisiología o Medicina en 1959 por este trabajo. La ADN polimerasa II fue descubierta por Thomas Kornberg (el hijo de Arthur Kornberg) y Malcolm E. Gefter en 1970 mientras aclaraba aún más el papel de Pol I en la replicación del ADN de E. coli. Se han encontrado tres ADN polimerasas más en E. coli, incluida la ADN polimerasa III (descubierta en la década de 1970) y las ADN polimerasas IV y V (descubiertas en 1999).
La función principal de la ADN polimerasa es sintetizar ADN a partir de desoxirribonucleótidos, los componentes básicos del ADN. Las copias de ADN se crean mediante el emparejamiento de nucleótidos con las bases presentes en cada hebra de la molécula de ADN original. Este emparejamiento siempre ocurre en combinaciones específicas, con citosina junto con guanina y timina junto con adenina, formando dos pares separados, respectivamente. Por el contrario, las polimerasas de ARN sintetizan ARN a partir de ribonucleótidos de ARN o ADN.
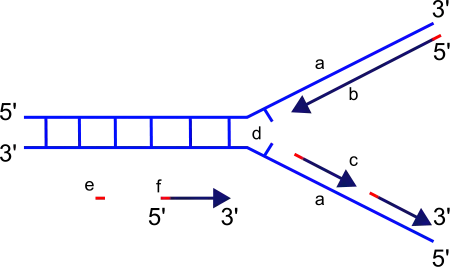
Al sintetizar ADN nuevo, la ADN polimerasa puede agregar nucleótidos libres solo al extremo 3 'de la cadena recién formada. Esto da como resultado el alargamiento de la hebra recién formada en una dirección de 5' a 3'.
Es importante tener en cuenta que la direccionalidad de la hebra recién formada (la hebra hija) es opuesta a la dirección en la que la ADN polimerasa se mueve a lo largo de la hebra molde. Dado que la ADN polimerasa requiere un grupo OH 3' libre para iniciar la síntesis, puede sintetizarse en una sola dirección extendiendo el extremo 3' de la cadena de nucleótidos preexistente. Por lo tanto, la ADN polimerasa se mueve a lo largo de la hebra molde en una dirección de 3' a 5', y la hebra hija se forma en una dirección de 5' a 3'. Esta diferencia permite que el ADN de doble cadena resultante formado esté compuesto por dos cadenas de ADN que son antiparalelas entre sí.
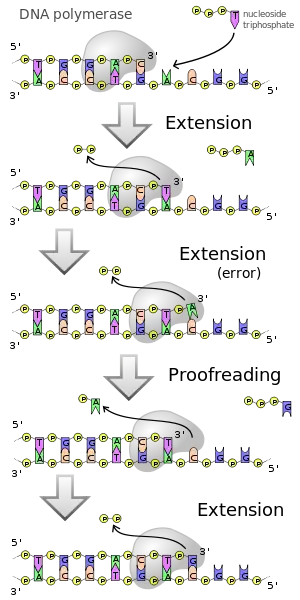
La función de la ADN polimerasa no es del todo perfecta, ya que la enzima comete un error por cada mil millones de pares de bases copiados. La corrección de errores es una propiedad de algunas, pero no de todas, las ADN polimerasas. Este proceso corrige errores en el ADN recién sintetizado. Cuando se reconoce un par de bases incorrecto, la ADN polimerasa retrocede un par de bases de ADN. La actividad de exonucleasa 3'–5' de la enzima permite eliminar el par de bases incorrecto (esta actividad se conoce como revisión). Después de la escisión de la base, la polimerasa puede reinsertar la base correcta y la replicación puede continuar. Esto preserva la integridad de la hebra de ADN original que se transmite a las células hijas.
La fidelidad es muy importante en la replicación del ADN. Los desajustes en el emparejamiento de bases de ADN pueden dar lugar a proteínas disfuncionales y podrían provocar cáncer. Muchas ADN polimerasas contienen un dominio de exonucleasa, que actúa en la detección de desajustes de pares de bases y actúa además en la eliminación del nucleótido incorrecto para ser reemplazado por el correcto.La forma y las interacciones que acomodan el par de bases de Watson y Crick son las que contribuyen principalmente a la detección o error. Los enlaces de hidrógeno juegan un papel clave en la unión e interacción de pares de bases. Se dice que la pérdida de una interacción, que ocurre en un desajuste, desencadena un cambio en el equilibrio, para la unión de la plantilla-cebador, de la polimerasa, al dominio exonucleasa. Además, la incorporación de un nucleótido equivocado provoca un retraso en la polimerización del ADN. Este retraso da tiempo para que el ADN cambie del sitio de la polimerasa al sitio de la exonucleasa. Diferentes cambios conformacionales y pérdida de interacción ocurren en diferentes desajustes. En un desajuste purina:pirimidina hay un desplazamiento de la pirimidina hacia el surco mayor y la purina hacia el surco menor. En relación con la forma de la ADN polimerasa'Los desajustes pirimidina:pirimidina y purina:purina presentan cambios menos notables ya que las bases se desplazan hacia el surco mayor y se experimenta menos impedimento estérico. Sin embargo, aunque los diferentes desajustes dan como resultado diferentes propiedades estéricas, la ADN polimerasa aún puede detectarlos y diferenciarlos de manera tan uniforme y mantener la fidelidad en la replicación del ADN. La polimerización del ADN también es crítica para muchos procesos de mutagénesis y se emplea ampliamente en biotecnologías.
Las polimerasas de ADN conocidas tienen una estructura muy conservada, lo que significa que sus subunidades catalíticas generales varían muy poco de una especie a otra, independientemente de las estructuras de sus dominios. Las estructuras conservadas suelen indicar funciones importantes e insustituibles de la célula, cuyo mantenimiento proporciona ventajas evolutivas. La forma se puede describir como similar a una mano derecha con dominios de pulgar, dedo y palma. El dominio palm parece funcionar al catalizar la transferencia de grupos fosforilo en la reacción de transferencia de fosforilo. El ADN se une a la palma cuando la enzima está activa. Se cree que esta reacción es catalizada por un mecanismo de dos iones metálicos. El dominio del dedo funciona para unir los trifosfatos de nucleósidos con la base molde. El dominio del pulgar juega un papel potencial en la procesividad, translocación,
La rápida catálisis de la ADN polimerasa se debe a su naturaleza procesiva. La procesividad es una característica de las enzimas que funcionan sobre sustratos poliméricos. En el caso de la ADN polimerasa, el grado de procesividad se refiere al número promedio de nucleótidos agregados cada vez que la enzima se une a una plantilla. La ADN polimerasa promedio requiere alrededor de un segundo para localizar y unir una unión cebador/plantilla. Una vez que se une, una polimerasa de ADN no procesante agrega nucleótidos a una velocidad de un nucleótido por segundo. Sin embargo, las polimerasas de ADN procesivas agregan múltiples nucleótidos por segundo, lo que aumenta drásticamente la tasa de síntesis de ADN. El grado de procesividad es directamente proporcional a la tasa de síntesis de ADN. La tasa de síntesis de ADN en una célula viva se determinó primero como la tasa de elongación del ADN del fago T4 en el fago infectado.E. coli. Durante el período de aumento exponencial del ADN a 37 °C, la tasa fue de 749 nucleótidos por segundo.
La capacidad de la ADN polimerasa para deslizarse a lo largo de la plantilla de ADN permite una mayor procesividad. Hay un aumento dramático en la procesividad en la bifurcación de replicación. Este aumento se ve facilitado por la asociación de la ADN polimerasa con proteínas conocidas como pinza deslizante de ADN. Las pinzas son múltiples subunidades proteicas asociadas en forma de anillo. Usando la hidrólisis de ATP, una clase de proteínas conocidas como proteínas de carga de abrazadera deslizante abren la estructura del anillo de las abrazaderas de ADN deslizantes que permiten la unión y la liberación de la hebra de ADN. La interacción proteína-proteína con la abrazadera evita que la ADN polimerasa se difunda desde la plantilla de ADN, lo que garantiza que la enzima se una a la misma unión cebador/plantilla y continúe la replicación.La ADN polimerasa cambia de conformación, aumentando la afinidad por la abrazadera cuando se asocia con ella y disminuyendo la afinidad cuando completa la replicación de un tramo de ADN para permitir la liberación de la abrazadera.
| ADN polimerasa familia A | |
|---|---|
 par c:o6-metil-guanina en la posición del par de bases de la polimerasa-2 par c:o6-metil-guanina en la posición del par de bases de la polimerasa-2 | |
| Identificadores | |
| Símbolo | ADN_pol_A |
| Pfam | PF00476 |
| interpro | IPR001098 |
| ELEGANTE | - |
| PROSITO | PDOC00412 |
| SCOP2 | 1 ppp / ALCANCE / SUPFAM |
| showEstructuras de proteínas disponibles: |
| ADN polimerasa familia B | |
|---|---|
 estructura cristalina de rb69 gp43 en complejo con adn que contiene timina glicol estructura cristalina de rb69 gp43 en complejo con adn que contiene timina glicol | |
| Identificadores | |
| Símbolo | ADN_pol_B |
| Pfam | PF00136 |
| clan pfam | CL0194 |
| interpro | IPR006134 |
| PROSITO | PDOC00107 |
| SCOP2 | 1noy / ALCANCE / SUPFAM |
| showEstructuras de proteínas disponibles: |
| ADN polimerasa tipo B, organelar y viral | |
|---|---|
 ADN polimerasa phi29, forma de cristal ortorrómbico, complejo ssdna ADN polimerasa phi29, forma de cristal ortorrómbico, complejo ssdna | |
| Identificadores | |
| Símbolo | ADN_pol_B_2 |
| Pfam | PF03175 |
| clan pfam | CL0194 |
| interpro | IPR004868 |
| showEstructuras de proteínas disponibles: |
Según la homología de secuencia, las ADN polimerasas se pueden subdividir en siete familias diferentes: A, B, C, D, X, Y y RT.
Algunos virus también codifican ADN polimerasas especiales, como la ADN polimerasa del virus de la hepatitis B. Estos pueden replicar selectivamente el ADN viral a través de una variedad de mecanismos. Los retrovirus codifican una polimerasa de ADN inusual llamada transcriptasa inversa, que es una polimerasa de ADN dependiente de ARN (RdDp). Polimeriza el ADN a partir de un molde de ARN. La terapia génica es la de los eucariotas. Euryarchaeota de PolD (heterodímero DP1/DP2|) eu telo
| Familia | Tipos de ADN polimerasa | Taxones | Ejemplos | Rasgo |
|---|---|---|---|---|
| UN | Polimerasas replicativas y reparadoras | eucariotas y procariotas | ADN polimerasa T7, Pol I, Pol γ, θ y ν | Dos dominios de exonucleasa (3'-5' y 5'-3') |
| B | Polimerasas replicativas y reparadoras | eucariotas y procariotas | Pol II, Pol B, Pol ζ, Pol α, δ y ε | 3'-5 exonucleasa (corrección); los virales usan cebador de proteína |
| C | Polimerasas replicativas | procariótico | Pol III | Exonucleasa 3'-5 (revisión) |
| D | Polimerasas replicativas | Euryarchaeota | PolD (heterodímero DP1/DP2) | Sin función de "mano", similar a la polimerasa de ARN de doble barril; Exonucleasa 3'-5 (revisión) |
| X | Polimerasas replicativas y reparadoras | eucariota | Pol β, Pol σ, Pol λ, Pol μ y desoxinucleotidil transferasa terminal | plantilla opcional; 5' fosfatasa (solo Pol β); función de "mano" débil |
| Y | Polimerasas replicativas y reparadoras | eucariotas y procariotas | Pol ι, Pol κ, Pol η, Pol IV y Pol V | Síntesis de translesión |
| RT | Polimerasas replicativas y reparadoras | Virus, retrovirus y eucariotas | Telomerasa, virus de la hepatitis B | dependiente de ARN |
Las polimerasas procarióticas existen en dos formas: polimerasa central y holoenzima. La polimerasa central sintetiza ADN a partir de la plantilla de ADN, pero no puede iniciar la síntesis sola o con precisión. La holoenzima inicia con precisión la síntesis.
Las polimerasas procariotas de la familia A incluyen la enzima ADN polimerasa I (Pol I), que está codificada por el gen polA y es ubicua entre los procariotas. Esta polimerasa reparadora participa en la reparación por escisión con actividad de exonucleasa 3'–5' y 5'–3' y el procesamiento de los fragmentos de Okazaki generados durante la síntesis de la cadena retardada. Pol I es la polimerasa más abundante y representa >95 % de la actividad polimerasa en E. coli; sin embargo, se han encontrado células que carecen de Pol I, lo que sugiere que la actividad de Pol I puede ser reemplazada por las otras cuatro polimerasas. Pol I agrega ~ 15-20 nucleótidos por segundo, mostrando así una pobre procesividad. En cambio, Pol I comienza a agregar nucleótidos en la unión cebador:plantilla de ARN conocida como origen de replicación (ori). Aproximadamente 400 pb aguas abajo del origen, la holoenzima Pol III se ensambla y se hace cargo de la replicación a una velocidad y naturaleza altamente procesadas.
Taq polimerasa es una enzima termoestable de esta familia que carece de capacidad de corrección.
La ADN polimerasa II es una polimerasa de la familia B codificada por el gen polB. Pol II tiene actividad de exonucleasa 3'–5' y participa en la reparación del ADN, el reinicio de la replicación para evitar lesiones, y su presencia celular puede saltar de ~30-50 copias por célula a ~200-300 durante la inducción de SOS. También se cree que Pol II es una copia de seguridad de Pol III, ya que puede interactuar con las proteínas holoenzimáticas y asumir un alto nivel de procesividad. Se cree que el papel principal de Pol II es la capacidad de dirigir la actividad de la polimerasa en la horquilla de replicación y ayudar a detener los desajustes terminales de derivación de Pol III.
La ADN polimerasa Pfu es una enzima estable al calor de esta familia que se encuentra en la arquea hipertermófila Pyrococcus furiosus. La clasificación detallada divide a la familia B en arqueas en B1, B2, B3, en la que B2 es un grupo de pseudoenzimas. Pfu pertenece a la familia B3. Otros PolB que se encuentran en arqueas son parte de "Casposons", transposones dependientes de Cas1. Algunos virus (incluida la ADN polimerasa Φ29) y los plásmidos mitocondriales también portan polB.
La holoenzima ADN polimerasa III es la principal enzima implicada en la replicación del ADN en E. coli y pertenece a la familia de polimerasas C. Consta de tres ensamblajes: el núcleo pol III, el factor de procesividad de abrazadera deslizante beta y el complejo de carga de abrazadera. El núcleo consta de tres subunidades: α, el centro de actividad de la polimerasa, ɛ, el corrector exonucleolítico y θ, que puede actuar como estabilizador de ɛ. El factor de procesividad de abrazadera deslizante beta también está presente por duplicado, uno para cada núcleo, para crear una abrazadera que encierra el ADN y permite una alta procesividad. El tercer conjunto es un complejo de cargador de abrazadera de siete subunidades (τ2γδδ′χψ).
El antiguo "modelo de trombón" de los libros de texto representa un complejo de elongación con dos equivalentes de la enzima central en cada horquilla de replicación (RF), uno para cada hebra, la rezagada y la líder. Sin embargo, la evidencia reciente de estudios de una sola molécula indica un promedio de tres equivalentes estequiométricos de enzima central en cada RF tanto para Pol III como para su contraparte en B. subtilis, PolC. La microscopía de fluorescencia en la célula ha revelado que la síntesis de la cadena principal puede no ser completamente continua y que la Pol III* (es decir, las subunidades de la holoenzima α, ε, τ, δ y χ sin la abrazadera deslizante ß2) tiene una alta frecuencia de disociación de la cadena activa. RF. En estos estudios, la tasa de rotación de la horquilla de replicación fue de aproximadamente 10 s para Pol III*, 47 s para la abrazadera deslizante ß2 y 15 m para la helicasa DnaB. Esto sugiere que la helicasa DnaB puede permanecer asociada de manera estable en los RF y servir como punto de nucleación para la holoenzima competente. Los estudios de una sola molécula in vitro han demostrado que Pol III* tiene una alta tasa de recambio de RF cuando está en exceso, pero permanece asociado de manera estable con las horquillas de replicación cuando la concentración es limitante. Otro estudio de una sola molécula mostró que la actividad helicasa de DnaB y el alargamiento de la hebra pueden proceder con una cinética estocástica desacoplada.
En E. coli, la ADN polimerasa IV (Pol IV) es una ADN polimerasa propensa a errores implicada en mutagénesis no dirigida. Pol IV es una polimerasa de la familia Y expresada por el gen dinB que se activa a través de la inducción de SOS causada por polimerasas estancadas en la horquilla de replicación. Durante la inducción de SOS, la producción de Pol IV aumenta diez veces y una de las funciones durante este tiempo es interferir con la procesividad de la holoenzima Pol III. Esto crea un punto de control, detiene la replicación y da tiempo para reparar las lesiones del ADN a través de la vía de reparación adecuada. Otra función de Pol IV es realizar la síntesis de translesión en la bifurcación de replicación estancada como, por ejemplo, eludir los aductos de N2-desoxiguanina a un ritmo más rápido que atravesar el ADN no dañado. Células que carecen deEl gen dinB tiene una mayor tasa de mutagénesis causada por agentes que dañan el ADN.
La ADN polimerasa V (Pol V) es una ADN polimerasa de la familia Y que participa en la respuesta SOS y en los mecanismos de reparación del ADN en la síntesis de translesiones. La transcripción de Pol V a través de los genes umuDC está altamente regulada para producir solo Pol V cuando el ADN dañado está presente en la célula generando una respuesta SOS. Las polimerasas estancadas hacen que RecA se una al ssDNA, lo que hace que la proteína LexA se autodigiera. LexA luego pierde su capacidad para reprimir la transcripción del operón umuDC. La misma nucleoproteína RecA-ssDNA modifica postraduccionalmente la proteína UmuD en proteína UmuD'. UmuD y UmuD' forman un heterodímero que interactúa con UmuC, que a su vez activa la actividad catalítica de la polimerasa de umuC en el ADN dañado. En E. coli, se ha propuesto un modelo de "cinturón de herramientas" de polimerasa para cambiar pol III con pol IV en una horquilla de replicación estancada, donde ambas polimerasas se unen simultáneamente a la abrazadera β. Sin embargo, aún no se ha demostrado en E. coli la participación de más de una polimerasa TLS trabajando en sucesión para eludir una lesión. Además, Pol IV puede catalizar tanto la inserción como la extensión con alta eficiencia, mientras que pol V se considera la principal polimerasa SOS TLS. Un ejemplo es la derivación del entrecruzamiento intracatenario de guanina y timina donde se demostró, sobre la base de la diferencia en las firmas mutacionales de las dos polimerasas, que la pol IV y la pol V compiten por el TLS del entrecruzamiento intracatenario.
En 1998, la familia D de ADN polimerasa fue descubierta en Pyrococcus furiosus y Methanococcus jannaschii. El complejo PolD es un heterodímero de dos cadenas, cada una codificada por DP1 (revisión pequeña) y DP2 (catalizador grande). A diferencia de otras ADN polimerasas, la estructura y el mecanismo del núcleo catalítico de DP2 se asemejan a las de las ARN polimerasas de múltiples subunidades. La interfaz DP1-DP2 se asemeja a la del dedo de zinc de la polimerasa eucariota de clase B y su pequeña subunidad. DP1, una exonucleasa similar a Mre11, es probablemente el precursor de la pequeña subunidad de Pol α y ε, lo que proporciona capacidades de corrección que ahora se pierden en los eucariotas. Su dominio HSH N-terminal es similar a las proteínas AAA, especialmente a la subunidad δ de Pol III y RuvB, en estructura.DP2 tiene un dominio KH Clase II. Pyrococcus abyssi polD es más estable al calor y más preciso que la polimerasa Taq, pero aún no se ha comercializado. Se ha propuesto que la ADN polimerasa de la familia D fue la primera en evolucionar en los organismos celulares y que la polimerasa replicativa del Último Ancestro Celular Universal (LUCA) pertenecía a la familia D.
Las polimerasas de la familia X contienen la conocida polimerasa eucariota pol β (beta), así como otras polimerasas eucariotas como Pol σ (sigma), Pol λ (lambda), Pol μ (mu) y desoxinucleotidil transferasa terminal (TdT). Las polimerasas de la familia X se encuentran principalmente en vertebrados y algunas se encuentran en plantas y hongos. Estas polimerasas tienen regiones altamente conservadas que incluyen dos motivos hélice-horquilla-hélice que son imprescindibles en las interacciones ADN-polimerasa. Un motivo está ubicado en el dominio de 8 kDa que interactúa con el ADN aguas abajo y un motivo está ubicado en el dominio del pulgar que interactúa con la hebra del cebador. Pol β, codificado por el gen POLB, es necesario para la reparación por escisión de base de parche corto, una vía de reparación de ADN que es esencial para reparar bases alquiladas u oxidadas, así como sitios abásicos. Pol λ y Pol μ, codificados por los genes POLL y POLM respectivamente, están involucrados en la unión de extremos no homólogos, un mecanismo para volver a unir roturas de doble cadena de ADN debido al peróxido de hidrógeno y la radiación ionizante, respectivamente. TdT se expresa solo en tejido linfoide y agrega "n nucleótidos" a las rupturas de doble cadena formadas durante la recombinación V(D)J para promover la diversidad inmunológica.
Pol α (alfa), Pol δ (delta) y Pol ε (épsilon) son miembros de las polimerasas de la familia B y son las principales polimerasas implicadas en la replicación del ADN nuclear. El complejo Pol α (complejo pol α-DNA primasa) consta de cuatro subunidades: la subunidad catalítica POLA1, la subunidad reguladora POLA2 y las subunidades de primasa pequeña y grande PRIM1 y PRIM2 respectivamente. Una vez que primasa ha creado el cebador de ARN, Pol α comienza la replicación alargando el cebador con ~ 20 nucleótidos. Debido a su alta procesividad, Pol δ se hace cargo de la síntesis de cadena principal y retrasada de Pol α. Pol δ es expresado por los genes POLD1, creando la subunidad catalítica, POLD2, POLD3 y POLD4 creando las otras subunidades que interactúan con el antígeno nuclear de células en proliferación (PCNA), que es una abrazadera de ADN que permite que Pol δ posea procesividad. Pol ε está codificado por el gen POLE1, la subunidad catalítica, POLE2 y POLE3. Se ha informado que la función de Pol ε es extender la cadena principal durante la replicación, mientras que Pol δ replica principalmente la cadena rezagada; sin embargo, la evidencia reciente sugirió que Pol δ también podría tener un papel en la replicación de la cadena principal de ADN. Se cree que la región "reliquia de polimerasa" del extremo C de Pol ε, a pesar de ser innecesaria para la actividad de la polimerasa, es esencial para la vitalidad celular. Se cree que la región C-terminal proporciona un punto de control antes de entrar en la anafase, brinda estabilidad a la holoenzima y agrega proteínas a la holoenzima necesarias para el inicio de la replicación. Pol ε tiene un dominio de "palma" más grande que proporciona una alta procesividad independientemente de PCNA.
En comparación con otras polimerasas de la Familia B, la familia de exonucleasas DEDD responsable de la corrección de pruebas está inactiva en Pol α. Pol ε es único porque tiene dos dominios de dedos de zinc y una copia inactiva de otra polimerasa de la familia B en su C-terminal. La presencia de este dedo de zinc tiene implicaciones en los orígenes de Eukaryota, que en este caso se ubica en el grupo Asgard con la polimerasa Archaeal B3.
Pol η (eta), Pol ι (iota) y Pol κ (kappa), son ADN polimerasas de la familia Y involucradas en la reparación del ADN mediante síntesis de traducción y codificadas por los genes POLH, POLI y POLK respectivamente. Los miembros de la familia Y tienen cinco motivos comunes para ayudar a unir el sustrato y el extremo del cebador y todos incluyen los dominios típicos del pulgar, la palma y los dedos de la mano derecha con dominios agregados como el dedo meñique (LF), el dominio asociado a la polimerasa (PAD) o pulsera. El sitio activo, sin embargo, difiere entre los miembros de la familia debido a las diferentes lesiones que se reparan. Las polimerasas de la familia Y son polimerasas de baja fidelidad, pero se ha demostrado que hacen más bien que mal, ya que las mutaciones que afectan a la polimerasa pueden causar diversas enfermedades, como el cáncer de piel y la variante xerodermia pigmentaria (XPS). La importancia de estas polimerasas se evidencia por el hecho de que el gen que codifica la ADN polimerasa η se conoce como XPV, porque la pérdida de este gen da como resultado la enfermedad Xeroderma Pigmentosum Variant. Pol η es particularmente importante para permitir la síntesis de translesión precisa del daño en el ADN resultante de la radiación ultravioleta. La funcionalidad de Pol κ no se comprende completamente, pero los investigadores han encontrado dos funciones probables. Se cree que Pol κ actúa como extensor o insertador de una base específica en ciertas lesiones del ADN. Las tres polimerasas de síntesis de translesión, junto con Rev1, se reclutan para las lesiones dañadas a través de polimerasas de ADN replicativas estancadas. Hay dos vías de reparación de daños que llevan a los investigadores a concluir que la vía elegida depende de qué hebra contiene el daño, la hebra principal o la rezagada.
Pol ζ, otra polimerasa de la familia B, está formada por dos subunidades Rev3, la subunidad catalítica, y Rev7 (MAD2L2), que aumenta la función catalítica de la polimerasa y participa en la síntesis de translesión. Pol ζ carece de actividad de exonucleasa de 3' a 5', es único en el sentido de que puede extender cebadores con desajustes terminales. Rev1 tiene tres regiones de interés en el dominio BRCT, el dominio de unión a ubiquitina y el dominio C-terminal y tiene la capacidad de transferasa de dCMP, que agrega desoxicitidina frente a las lesiones que detendrían las polimerasas replicativas Pol δ y Pol ε. Estas polimerasas estancadas activan complejos de ubiquitina que a su vez disocian las polimerasas de replicación y reclutan Pol ζ y Rev1. Juntos, Pol ζ y Rev1 agregan desoxicitidina y Pol ζ se extiende más allá de la lesión. A través de un proceso aún no determinado, Pol ζ se disocia y las polimerasas de replicación se vuelven a asociar y continúan la replicación. Pol ζ y Rev1 no son necesarios para la replicación, pero la pérdida del gen REV3 en la levadura en ciernes puede causar una mayor sensibilidad a los agentes que dañan el ADN debido al colapso de las horquillas de replicación donde se han estancado las polimerasas de replicación.
La telomerasa es una ribonucleoproteína que funciona para replicar los extremos de los cromosomas lineales, ya que la ADN polimerasa normal no puede replicar los extremos o telómeros. El saliente 3' monocatenario del cromosoma bicatenario con la secuencia 5'-TTAGGG-3' recluta la telomerasa. La telomerasa actúa como otras ADN polimerasas extendiendo el extremo 3' pero, a diferencia de otras ADN polimerasas, la telomerasa no requiere una plantilla. La subunidad TERT, un ejemplo de transcriptasa inversa, utiliza la subunidad de ARN para formar la unión cebador-plantilla que permite que la telomerasa extienda el extremo 3' de los extremos cromosómicos. Se cree que la disminución gradual del tamaño de los telómeros como resultado de muchas replicaciones a lo largo de la vida está asociada con los efectos del envejecimiento.
Pol γ (gamma), Pol θ (theta) y Pol ν (nu) son polimerasas de la familia A. Durante mucho tiempo se pensó que Pol γ, codificada por el gen POLG, era la única polimerasa mitocondrial. Sin embargo, investigaciones recientes muestran que al menos Pol β (beta), una polimerasa de la familia X, también está presente en las mitocondrias. Cualquier mutación que conduzca a Pol γ limitada o no funcional tiene un efecto significativo en el mtDNA y es la causa más común de trastornos mitocondriales heredados autosómicos. Pol γ contiene un dominio de polimerasa C-terminal y un dominio de exonucleasa 3'–5' N-terminal que están conectados a través de la región enlazadora, que se une a la subunidad accesoria. La subunidad accesoria se une al ADN y es necesaria para la procesividad de Pol γ. La mutación puntual A467T en la región enlazadora es responsable de más de un tercio de todos los trastornos mitocondriales asociados con Pol γ. Si bien muchos homólogos de Pol θ, codificados por el gen POLQ, se encuentran en eucariotas, su función no se comprende claramente. La secuencia de aminoácidos en el extremo C es lo que clasifica a Pol θ como polimerasa de la familia A, aunque la tasa de error de Pol θ está más estrechamente relacionada con las polimerasas de la familia Y. Pol θ extiende los extremos del cebador que no coinciden y puede pasar por alto los sitios básicos agregando un nucleótido. También tiene actividad de desoxirribofosfodiesterasa (dRPasa) en el dominio de la polimerasa y puede mostrar actividad de ATPasa muy cerca de ssDNA. Se considera que Pol ν (nu) es la menos eficaz de las enzimas polimerasas. Sin embargo, la ADN polimerasa nu juega un papel activo en la reparación de la homología durante las respuestas celulares a los entrecruzamientos, cumpliendo su función en un complejo con la helicasa.
Las plantas usan dos polimerasas de la Familia A para copiar los genomas mitocondrial y plástido. Son más similares a la Pol I bacteriana que a la Pol γ de los mamíferos.
Los retrovirus codifican una polimerasa de ADN inusual llamada transcriptasa inversa, que es una polimerasa de ADN dependiente de ARN (RdDp) que sintetiza ADN a partir de una plantilla de ARN. La familia de las transcriptasas inversas contiene tanto la funcionalidad de la ADN polimerasa como la funcionalidad de la ARNasa H, que degrada el ARN apareado en base al ADN. Un ejemplo de un retrovirus es el VIH. La transcriptasa inversa se emplea comúnmente en la amplificación de ARN con fines de investigación. Usando una plantilla de ARN, la PCR puede utilizar la transcriptasa inversa, creando una plantilla de ADN. Esta nueva plantilla de ADN se puede utilizar para la amplificación por PCR típica. Los productos de tal experimento son, por lo tanto, productos de PCR amplificados a partir de ARN.
Cada partícula de retrovirus del VIH contiene dos genomas de ARN, pero, después de una infección, cada virus genera solo un provirus. Después de la infección, la transcripción inversa se acompaña de un cambio de plantilla entre las dos copias del genoma (recombinación de elección de copia). En cada ciclo de replicación ocurren de 5 a 14 eventos de recombinación por genoma. El cambio de plantilla (recombinación) parece ser necesario para mantener la integridad del genoma y como mecanismo de reparación para salvar genomas dañados.
El bacteriófago (fago) T4 codifica una ADN polimerasa que cataliza la síntesis de ADN en una dirección de 5' a 3'. La polimerasa del fago también tiene una actividad de exonucleasa que actúa en una dirección de 3' a 5', y esta actividad se emplea en la revisión y edición de bases recién insertadas. Se observó que un fago mutante con una polimerasa de ADN sensible a la temperatura, cuando se cultivaba a temperaturas permisivas, experimentaba una recombinación a frecuencias que eran aproximadamente dos veces más altas que las del fago de tipo salvaje.
Se propuso que una alteración mutacional en la ADN polimerasa del fago puede estimular el cambio de cadena de plantilla (recombinación de elección de copia) durante la replicación.
(leer más)
El dogma central de la biología molecular es una explicación del flujo de información genética dentro de un sistema biológico. A menudo se dice que el... (leer más)
Una vacuola es un orgánulo unido a una membrana que está presente en las células vegetales y fúngicas y en algunas células protistas, animales y... (leer más)