RNA mensageiro
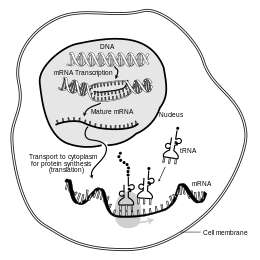
Na biologia molecular, o ácido ribonucléico mensageiro (mRNA) é uma molécula de RNA de cadeia simples que corresponde à sequência genética de um gene e é lida por um ribossomo no processo de síntese de uma proteína.
O mRNA é criado durante o processo de transcrição, onde uma enzima (RNA polimerase) converte o gene em mRNA transcrito primário (também conhecido como pré-mRNA). Esse pré-mRNA geralmente ainda contém íntrons, regiões que não irão codificar a sequência final de aminoácidos. Estes são removidos no processo de splicing do RNA, deixando apenas os éxons, regiões que irão codificar a proteína. Esta sequência de exon constitui mRNA maduro. O mRNA maduro é então lido pelo ribossomo e, utilizando aminoácidos transportados pelo RNA de transferência (tRNA), o ribossomo cria a proteína. Este processo é conhecido como tradução. Todos esses processos fazem parte do dogma central da biologia molecular, que descreve o fluxo da informação genética em um sistema biológico.
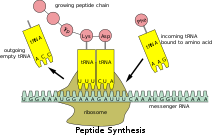
Assim como no DNA, a informação genética no mRNA está contida na sequência de nucleotídeos, que são arranjados em códons que consistem em três ribonucleotídeos cada. Cada códon codifica um aminoácido específico, exceto os códons de parada, que terminam a síntese de proteínas. A tradução de códons em aminoácidos requer dois outros tipos de RNA: o RNA de transferência, que reconhece o códon e fornece o aminoácido correspondente, e o RNA ribossômico (rRNA), o componente central da maquinaria de fabricação de proteínas do ribossomo.
O conceito de mRNA foi desenvolvido por Sydney Brenner e Francis Crick em 1960 durante uma conversa com François Jacob. Em 1961, o mRNA foi identificado e descrito independentemente por uma equipe formada por Brenner, Jacob e Matthew Meselson, e outra equipe liderada por James Watson. Ao analisar os dados em preparação para publicação, Jacob e Jacques Monod cunharam o nome "RNA mensageiro".
Síntese
A breve existência de uma molécula de mRNA começa com a transcrição e, por fim, termina com a degradação. Durante sua vida, uma molécula de mRNA também pode ser processada, editada e transportada antes da tradução. Moléculas de mRNA eucarióticas geralmente requerem processamento e transporte extensivos, enquanto moléculas de mRNA procarióticas não. Uma molécula de mRNA eucariótica e as proteínas que a cercam são chamadas de RNP mensageiro.
Transcrição
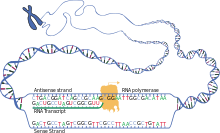
A transcrição é quando o RNA é copiado do DNA. Durante a transcrição, a RNA polimerase faz uma cópia de um gene do DNA para o mRNA conforme necessário. Este processo difere ligeiramente em eucariotos e procariotos. Uma diferença notável é que a polimerase de RNA procariótica se associa a enzimas de processamento de DNA durante a transcrição, de modo que o processamento pode prosseguir durante a transcrição. Portanto, isso faz com que a nova fita de mRNA se torne uma fita dupla, produzindo uma fita complementar conhecida como fita de tRNA, que quando combinadas são incapazes de formar estruturas a partir do pareamento de bases. Além disso, o molde para o mRNA é a fita complementar do tRNA, que é idêntica em sequência à sequência do anticódon à qual o DNA se liga. O produto de vida curta, não processado ou parcialmente processado é denominado mRNA precursor ou pré-mRNA; uma vez completamente processado, é denominado mRNA maduro.
Processamento de pré-mRNA eucariótico
O processamento do mRNA difere muito entre eucariotos, bactérias e archaea. O mRNA não eucariótico é, em essência, maduro após a transcrição e não requer processamento, exceto em casos raros. O pré-mRNA eucariótico, no entanto, requer várias etapas de processamento antes de seu transporte para o citoplasma e sua tradução pelo ribossomo.
Emenda
O processamento extensivo do pré-mRNA eucariótico que leva ao mRNA maduro é o splicing do RNA, um mecanismo pelo qual íntrons ou outrons (regiões não codificantes) são removidos e éxons (regiões codificantes) são unidos.
5' adição de limite
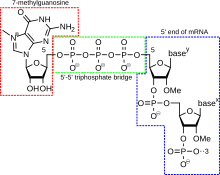
A 5' cap (também denominado cap de RNA, cap de RNA 7-metilguanosina ou cap de RNA m7G) é um nucleotídeo de guanina modificado que foi adicionado à "frente& #34; ou 5' final de um RNA mensageiro eucariótico logo após o início da transcrição. O 5' cap consiste em um resíduo terminal de 7-metilguanosina que está ligado através de uma ligação 5'-5'-trifosfato ao primeiro nucleotídeo transcrito. Sua presença é crítica para o reconhecimento pelo ribossomo e proteção contra RNases.
A adição de cap é acoplada à transcrição e ocorre co-transcricionalmente, de modo que cada um influencia o outro. Logo após o início da transcrição, o 5' A extremidade do mRNA que está sendo sintetizado é ligada por um complexo cap-sintetizador associado à RNA polimerase. Este complexo enzimático catalisa as reações químicas necessárias para o capeamento do mRNA. A síntese ocorre como uma reação bioquímica em várias etapas.
Edição
Em alguns casos, um mRNA será editado, alterando a composição de nucleotídeos desse mRNA. Um exemplo em humanos é o mRNA da apolipoproteína B, que é editado em alguns tecidos, mas não em outros. A edição cria um códon de parada inicial, que, após a tradução, produz uma proteína mais curta.
Poliadenilação
A poliadenilação é a ligação covalente de uma porção poliadenilila a uma molécula de RNA mensageiro. Em organismos eucarióticos, a maioria das moléculas de RNA mensageiro (mRNA) são poliadeniladas na região 3' final, mas estudos recentes mostraram que curtos períodos de uridina (oligouridilação) também são comuns. A cauda poli(A) e a proteína ligada a ela ajudam a proteger o mRNA da degradação por exonucleases. A poliadenilação também é importante para a terminação da transcrição, exportação do mRNA do núcleo e tradução. O mRNA também pode ser poliadenilado em organismos procarióticos, onde as caudas poli(A) atuam para facilitar, em vez de impedir, a degradação exonucleolítica.
A poliadenilação ocorre durante e/ou imediatamente após a transcrição do DNA em RNA. Após o término da transcrição, a cadeia de mRNA é clivada pela ação de um complexo de endonuclease associado à RNA polimerase. Após a clivagem do mRNA, cerca de 250 resíduos de adenosina são adicionados ao 3' termina no local de clivagem. Esta reação é catalisada pela poliadenilato polimerase. Assim como no splicing alternativo, pode haver mais de uma variante de poliadenilação de um mRNA.
Também ocorrem mutações no local de poliadenilação. O transcrito de RNA primário de um gene é clivado no local de adição poli-A, e 100-200 A's são adicionados ao 3' final do ARN. Se este local for alterado, uma construção de mRNA anormalmente longa e instável será formada.
Transporte
Outra diferença entre eucariotos e procariontes é o transporte de mRNA. Como a transcrição e a tradução eucarióticas são compartimentalmente separadas, os mRNAs eucarióticos devem ser exportados do núcleo para o citoplasma - um processo que pode ser regulado por diferentes vias de sinalização. Os mRNAs maduros são reconhecidos por suas modificações processadas e então exportados através do poro nuclear por ligação às proteínas cap-binding CBP20 e CBP80, bem como ao complexo de transcrição/exportação (TREX). Múltiplas vias de exportação de mRNA foram identificadas em eucariotos.
Em células espacialmente complexas, alguns mRNAs são transportados para destinos subcelulares específicos. Em neurônios maduros, certos mRNA são transportados do soma para os dendritos. Um local de tradução do mRNA é nos polirribossomos localizados seletivamente abaixo das sinapses. O mRNA para Arc/Arg3.1 é induzido por atividade sináptica e localiza-se seletivamente perto de sinapses ativas com base em sinais gerados por receptores NMDA. Outros mRNAs também se movem para os dendritos em resposta a estímulos externos, como o mRNA de β-actina. Após a exportação do núcleo, o mRNA da actina se associa ao ZBP1 e à subunidade 40S. O complexo é ligado por uma proteína motora e é transportado para o local alvo (extensão da neurite) ao longo do citoesqueleto. Eventualmente ZBP1 é fosforilado por Src para que a tradução seja iniciada. Nos neurônios em desenvolvimento, os mRNAs também são transportados para os axônios em crescimento e, especialmente, para os cones de crescimento. Muitos mRNAs são marcados com os chamados "códigos postais" que direcionam seu transporte para um local específico. Os mRNAs também podem ser transferidos entre células de mamíferos através de estruturas chamadas nanotubos de tunelamento.
Tradução
Como o mRNA procariótico não precisa ser processado ou transportado, a tradução pelo ribossomo pode começar imediatamente após o término da transcrição. Portanto, pode-se dizer que a tradução procariótica está acoplada à transcrição e ocorre co-transcricionalmente.
O mRNA eucariótico que foi processado e transportado para o citoplasma (ou seja, mRNA maduro) pode então ser traduzido pelo ribossomo. A tradução pode ocorrer nos ribossomos flutuando livremente no citoplasma ou direcionada ao retículo endoplasmático pela partícula de reconhecimento de sinal. Portanto, ao contrário dos procariotos, a tradução eucariótica não está diretamente acoplada à transcrição. É até possível, em alguns contextos, que os níveis reduzidos de mRNA sejam acompanhados por níveis aumentados de proteína, como foi observado para os níveis de mRNA/proteína de EEF1A1 no câncer de mama.
Estrutura
Regiões de codificação
As regiões codificadoras são compostas por códons, que são decodificados e traduzidos em proteínas pelo ribossomo; em eucariotos geralmente em um e em procariotos geralmente em vários. As regiões de codificação começam com o códon de início e terminam com um códon de parada. Em geral, o códon de início é um tripleto AUG e o códon de parada é UAG ("amber"), UAA ("ochre") ou UGA ("opala"). As regiões codificantes tendem a ser estabilizadas por pares de bases internos; isso impede a degradação. Além de codificarem proteínas, porções de regiões codificadoras podem servir como sequências reguladoras no pré-mRNA como intensificadores de splicing exônico ou silenciadores de splicing exônico.
Regiões não traduzidas
Regiões não traduzidas (UTRs) são seções do mRNA antes do códon de início e após o códon de parada que não são traduzidas, denominadas região não traduzida de cinco primos (5' UTR) e região de três primos não traduzidos (3' UTR), respectivamente. Essas regiões são transcritas com a região de codificação e, portanto, são exônicas, pois estão presentes no mRNA maduro. Vários papéis na expressão gênica foram atribuídos às regiões não traduzidas, incluindo estabilidade do mRNA, localização do mRNA e eficiência da tradução. A capacidade de um UTR de realizar essas funções depende da sequência do UTR e pode diferir entre os mRNAs. Variantes genéticas em 3' UTR também foram implicados na suscetibilidade a doenças por causa da mudança na estrutura do RNA e na tradução de proteínas.
A estabilidade dos mRNAs pode ser controlada pelo 5' UTR e/ou 3' UTR devido à afinidade variável por enzimas que degradam o RNA chamadas ribonucleases e por proteínas auxiliares que podem promover ou inibir a degradação do RNA. (Veja também elemento de estabilidade rico em C.)
A eficiência da tradução, incluindo às vezes a inibição completa da tradução, pode ser controlada pelos UTRs. Proteínas que se ligam a 3' ou 5' UTR pode afetar a tradução influenciando a capacidade do ribossomo de se ligar ao mRNA. MicroRNAs ligados ao 3' A UTR também pode afetar a eficiência da tradução ou a estabilidade do mRNA.
Pensa-se que a localização citoplasmática do mRNA seja uma função do 3' UTR. As proteínas que são necessárias em uma determinada região da célula também podem ser traduzidas para lá; nesse caso, o 3' UTR pode conter sequências que permitem que o transcrito seja localizado nesta região para tradução.
Alguns dos elementos contidos em regiões não traduzidas formam uma estrutura secundária característica quando transcritos em RNA. Esses elementos estruturais de mRNA estão envolvidos na regulação do mRNA. Alguns, como o elemento SECIS, são alvos para as proteínas se ligarem. Uma classe de elemento de mRNA, os riboswitches, ligam diretamente pequenas moléculas, alterando sua dobra para modificar os níveis de transcrição ou tradução. Nesses casos, o mRNA se auto-regula.
Cauda poli(A)
Os 3' A cauda poli(A) é uma longa sequência de nucleotídeos de adenina (muitas vezes várias centenas) adicionados à cadeia 3' final do pré-mRNA. Essa cauda promove a exportação do núcleo e a tradução e protege o mRNA da degradação.
ARNm monocistrônico vs policistrônico
Diz-se que uma molécula de mRNA é monocistrônica quando contém a informação genética para traduzir apenas uma única cadeia de proteína (polipeptídeo). Este é o caso da maioria dos mRNAs eucarióticos. Por outro lado, o mRNA policistrônico carrega vários quadros de leitura aberta (ORFs), cada um dos quais é traduzido em um polipeptídeo. Esses polipeptídeos geralmente têm uma função relacionada (muitas vezes são as subunidades que compõem uma proteína complexa final) e sua sequência de codificação é agrupada e regulada em uma região reguladora, contendo um promotor e um operador. A maior parte do mRNA encontrado em bactérias e archaea é policistrônico, assim como o genoma mitocondrial humano. O mRNA dicistrônico ou bicistrônico codifica apenas duas proteínas.
Circularização do MRNA
Em eucariotos, as moléculas de mRNA formam estruturas circulares devido a uma interação entre o eIF4E e a proteína de ligação poli(A), que se ligam ao eIF4G, formando uma ponte mRNA-proteína-mRNA. Acredita-se que a circularização promova a ciclagem de ribossomos no mRNA, levando à tradução com eficiência de tempo e também pode funcionar para garantir que apenas o mRNA intacto seja traduzido (o mRNA parcialmente degradado caracteristicamente não possui tampa m7G ou cauda poli-A).
Existem outros mecanismos de circularização, particularmente no mRNA do vírus. O mRNA do poliovírus usa uma seção de trevo em direção ao seu 5' final para se ligar a PCBP2, que se liga à proteína de ligação poli(A), formando o conhecido círculo mRNA-proteína-mRNA. Barley yellow dwarf virus tem ligação entre os segmentos de mRNA em seu 5' fim e 3' final (chamado de loops de haste de beijo), circularizando o mRNA sem nenhuma proteína envolvida.
Os genomas de vírus de RNA (cujas cadeias + são traduzidas como mRNA) também são comumente circularizados. Durante a replicação do genoma, a circularização atua para aumentar as velocidades de replicação do genoma, ciclando a polimerase de RNA dependente de RNA viral da mesma forma que o ribossomo é hipotetizado para o ciclo.
Degradação
Diferentes mRNAs dentro da mesma célula têm tempos de vida distintos (estabilidades). Em células bacterianas, mRNAs individuais podem sobreviver de segundos a mais de uma hora. No entanto, o tempo de vida é em média entre 1 e 3 minutos, tornando o mRNA bacteriano muito menos estável do que o mRNA eucariótico. Em células de mamíferos, o tempo de vida do mRNA varia de alguns minutos a dias. Quanto maior a estabilidade de um mRNA, mais proteína pode ser produzida a partir desse mRNA. O tempo de vida limitado do mRNA permite que uma célula altere rapidamente a síntese de proteínas em resposta às suas necessidades de mudança. Existem muitos mecanismos que levam à destruição de um mRNA, alguns dos quais são descritos a seguir.
Degradação do mRNA procariótico
Em geral, em procariotos, o tempo de vida do mRNA é muito menor do que em eucariotos. Os procariotos degradam as mensagens usando uma combinação de ribonucleases, incluindo endonucleases, 3' exonucleases e 5' exonucleases. Em alguns casos, pequenas moléculas de RNA (sRNA) com dezenas a centenas de nucleotídeos de comprimento podem estimular a degradação de mRNAs específicos por pareamento de bases com sequências complementares e facilitando a clivagem da ribonuclease pela RNase III. Foi recentemente demonstrado que as bactérias também têm uma espécie de 5' cap consistindo de um trifosfato no 5' fim. A remoção de dois dos fosfatos deixa um 5' monofosfato, fazendo com que a mensagem seja destruída pela exonuclease RNase J, que degrada 5' para 3'.
Turnance de mRNA eucariótico
Dentro das células eucarióticas, existe um equilíbrio entre os processos de tradução e decaimento do mRNA. As mensagens que estão sendo traduzidas ativamente são ligadas por ribossomos, os fatores de iniciação eucariótica eIF-4E e eIF-4G e a proteína de ligação poli(A). eIF-4E e eIF-4G bloqueiam a enzima decapante (DCP2), e a proteína de ligação a poli(A) bloqueia o complexo do exossomo, protegendo as extremidades da mensagem. O equilíbrio entre tradução e decaimento é refletido no tamanho e abundância de estruturas citoplasmáticas conhecidas como P-corpos. A cauda poli(A) do mRNA é encurtada por exonucleases especializadas que são direcionadas a RNAs mensageiros específicos por uma combinação de sequências reguladoras cis no RNA e proteínas de ligação ao RNA de ação trans. Acredita-se que a remoção da cauda de Poly(A) interrompa a estrutura circular da mensagem e desestabilize o complexo de ligação do cap. A mensagem é então sujeita a degradação pelo complexo exossomal ou pelo complexo de decapagem. Dessa forma, as mensagens inativas podem ser destruídas rapidamente, enquanto as mensagens ativas permanecem intactas. O mecanismo pelo qual a tradução para e a mensagem é transferida para complexos de decaimento não é compreendido em detalhes.
Decadência do elemento rico em AU
A presença de elementos ricos em AU em alguns mRNAs de mamíferos tende a desestabilizar esses transcritos por meio da ação de proteínas celulares que se ligam a essas sequências e estimulam a remoção da cauda de poli(A). Acredita-se que a perda da cauda poli(A) promova a degradação do mRNA ao facilitar o ataque tanto pelo complexo do exossomo quanto pelo complexo de decapagem. A rápida degradação do mRNA por meio de elementos ricos em AU é um mecanismo crítico para prevenir a superprodução de potentes citocinas, como o fator de necrose tumoral (TNF) e o fator estimulador de colônias de macrófagos e granulócitos (GM-CSF). Elementos ricos em AU também regulam a biossíntese de fatores de transcrição proto-oncogênicos como c-Jun e c-Fos.
Deterioração mediada por absurdos
Mensagens eucarióticas estão sujeitas a vigilância por decaimento mediado por nonsense (NMD), que verifica a presença de códons de parada prematuros (códons sem sentido) na mensagem. Estes podem surgir por meio de splicing incompleto, recombinação V(D)J no sistema imunológico adaptativo, mutações no DNA, erros de transcrição, varredura com vazamento pelo ribossomo causando uma mudança de quadro e outras causas. A detecção de um códon de parada prematuro desencadeia a degradação do mRNA em 5' decapagem, 3' remoção da cauda poli(A) ou clivagem endonucleolítica.
Pequeno RNA de interferência (siRNA)
Em metazoários, pequenos RNAs interferentes (siRNAs) processados por Dicer são incorporados a um complexo conhecido como complexo silenciador induzido por RNA ou RISC. Este complexo contém uma endonuclease que cliva mensagens perfeitamente complementares às quais o siRNA se liga. Os fragmentos de mRNA resultantes são então destruídos por exonucleases. siRNA é comumente usado em laboratórios para bloquear a função de genes em cultura de células. Acredita-se que faça parte do sistema imunológico inato como uma defesa contra vírus de RNA de fita dupla.
MicroRNA (miRNA)
MicroRNAs (miRNAs) são pequenos RNAs que normalmente são parcialmente complementares a sequências em RNAs mensageiros de metazoários. A ligação de um miRNA a uma mensagem pode reprimir a tradução dessa mensagem e acelerar a remoção da cauda poli(A), acelerando assim a degradação do mRNA. O mecanismo de ação dos miRNAs é objeto de intensa pesquisa.
Outros mecanismos de decaimento
Existem outras maneiras pelas quais as mensagens podem ser degradadas, incluindo decaimento ininterrupto e silenciamento por RNA de interação com Piwi (piRNA), entre outros.
Aplicativos
A administração de uma sequência de RNA mensageiro modificado por nucleosídeo pode fazer com que uma célula produza uma proteína, que por sua vez pode tratar diretamente uma doença ou funcionar como uma vacina; mais indiretamente, a proteína poderia levar uma célula-tronco endógena a se diferenciar da maneira desejada.
Os principais desafios da terapia de RNA centram-se na entrega do RNA às células apropriadas. Os desafios incluem o fato de que as sequências de RNA nu naturalmente se degradam após a preparação; eles podem acionar o sistema imunológico do corpo para atacá-los como invasores; e são impermeáveis à membrana celular. Uma vez dentro da célula, eles devem deixar o mecanismo de transporte da célula para agir dentro do citoplasma, que abriga os ribossomos necessários.
Superando esses desafios, o mRNA como terapêutico foi apresentado pela primeira vez em 1989 "após o desenvolvimento de uma técnica de transfecção in vitro amplamente aplicável." Na década de 1990, vacinas de mRNA para câncer personalizado foram desenvolvidas, contando com mRNA não modificado por nucleosídeo. As terapias baseadas em mRNA continuam a ser investigadas como um método de tratamento ou terapia tanto para o câncer quanto para doenças inflamatórias autoimunes, metabólicas e respiratórias. Terapias de edição de genes, como CRISPR, também podem se beneficiar do uso de mRNA para induzir as células a produzir a proteína Cas desejada.
Desde a década de 2010, as vacinas de RNA e outras terapias de RNA são consideradas "uma nova classe de medicamentos" As primeiras vacinas baseadas em mRNA receberam autorização restrita e foram lançadas em todo o mundo durante a pandemia de COVID-19 pela vacina Pfizer–BioNTech COVID-19 e Moderna, por exemplo.
História
Vários estudos de biologia molecular durante a década de 1950 indicaram que o RNA desempenhava algum tipo de papel na síntese de proteínas, mas esse papel não era claramente compreendido. Por exemplo, em um dos primeiros relatórios, Jacques Monod e sua equipe mostraram que a síntese de RNA era necessária para a síntese de proteínas, especificamente durante a produção da enzima β-galactosidase na bactéria E. coli. Arthur Pardee também encontrou acúmulo de RNA semelhante em 1954. Em 1953, Alfred Hershey, June Dixon e Martha Chase descreveram um certo DNA contendo citosina (indicando que era RNA) que desapareceu rapidamente após sua síntese em E. coli. Em retrospectiva, esta pode ter sido uma das primeiras observações da existência de mRNA, mas não foi reconhecida na época como tal.
A ideia do mRNA foi concebida pela primeira vez por Sydney Brenner e Francis Crick em 15 de abril de 1960 no King's College, Cambridge, enquanto François Jacob lhes contava sobre um experimento recente conduzido pelo próprio Arthur Pardee e Monod (o chamado experimento PaJaMo, que não provou a existência do mRNA, mas sugeriu a possibilidade de sua existência). Com o incentivo de Crick, Brenner e Jacob imediatamente começaram a testar essa nova hipótese e contataram Matthew Meselson, do Instituto de Tecnologia da Califórnia, para obter assistência. Durante o verão de 1960, Brenner, Jacob e Meselson conduziram um experimento no laboratório de Meselson na Caltech, que foi o primeiro a provar a existência do mRNA. Naquele outono, Jacob e Monod cunharam o nome "RNA mensageiro" e desenvolveu o primeiro quadro teórico para explicar a sua função.
Em fevereiro de 1961, James Watson revelou que seu grupo de pesquisa baseado em Harvard estava logo atrás deles com uma série de experimentos cujos resultados apontavam aproximadamente na mesma direção. Brenner e os outros concordaram com o pedido de Watson para adiar a publicação de suas descobertas de pesquisa. Como resultado, os artigos de Brenner e Watson foram publicados simultaneamente na mesma edição da Nature em maio de 1961, enquanto no mesmo mês, Jacob e Monod publicaram sua estrutura teórica para mRNA no Journal of Biologia Molecular.
Contenido relacionado
DNA ligase
Kary Mullis
Adenilil ciclase
Eletroforese em gel
Enzima