Replicação do DNA
Na biologia molecular, replicação de DNA é o processo biológico de produzir duas réplicas idênticas de DNA de uma molécula de DNA original. A replicação de DNA ocorre em todos os organismos vivos atuando como a parte mais essencial para a herança biológica. Isto é essencial para a divisão celular durante o crescimento e reparação de tecidos danificados, enquanto também garante que cada uma das novas células recebe sua própria cópia do DNA. A célula possui a propriedade distintiva da divisão, o que torna a replicação do DNA essencial.
O DNA é formado por uma dupla hélice de duas fitas complementares. A dupla hélice descreve a aparência de um DNA de fita dupla que é, portanto, composto de duas fitas lineares que correm opostas uma à outra e se torcem juntas para formar. Durante a replicação, essas fitas são separadas. Cada fita da molécula de DNA original serve então como modelo para a produção de sua contraparte, um processo conhecido como replicação semiconservativa. Como resultado da replicação semiconservativa, a nova hélice será composta de uma fita de DNA original, bem como de uma fita recém-sintetizada. Mecanismos celulares de revisão e verificação de erros garantem uma fidelidade quase perfeita para a replicação do DNA.
Em uma célula, a replicação do DNA começa em locais específicos, ou origens de replicação, no genoma que contém o material genético de um organismo. O desenrolamento do DNA na origem e a síntese de novos filamentos, acomodados por uma enzima conhecida como helicase, resulta em forquilhas de replicação crescendo bidirecionalmente a partir da origem. Várias proteínas estão associadas à forquilha de replicação para ajudar no início e na continuação da síntese de DNA. Mais proeminentemente, a DNA polimerase sintetiza as novas fitas adicionando nucleotídeos que complementam cada fita (modelo). A replicação do DNA ocorre durante o estágio S da interfase.
A replicação do DNA (amplificação do DNA) também pode ser realizada in vitro (artificialmente, fora de uma célula). Polimerases de DNA isoladas de células e iniciadores de DNA artificiais podem ser usadas para iniciar a síntese de DNA em sequências conhecidas em uma molécula de DNA modelo. A reação em cadeia da polimerase (PCR), a reação em cadeia da ligase (LCR) e a amplificação mediada por transcrição (TMA) são exemplos. Em março de 2021, pesquisadores relataram evidências sugerindo que uma forma preliminar de RNA de transferência, um componente necessário da tradução, a síntese biológica de novas proteínas de acordo com o código genético, poderia ter sido uma molécula replicadora no início do desenvolvimento da vida. ou abiogênese.
Estrutura do DNA
O DNA existe como uma estrutura de fita dupla, com ambas as fitas enroladas juntas para formar a dupla hélice característica. Cada fita única de DNA é uma cadeia de quatro tipos de nucleotídeos. Os nucleotídeos no DNA contêm um açúcar desoxirribose, um fosfato e uma nucleobase. Os quatro tipos de nucleotídeos correspondem às quatro nucleobases adenina, citosina, guanina e timina, comumente abreviadas como A, C, G e T. Adenina e guanina são bases purinas, enquanto citosina e timina são pirimidinas. Esses nucleotídeos formam ligações fosfodiéster, criando o esqueleto fosfato-desoxirribose da dupla hélice do DNA com as nucleobases apontando para dentro (ou seja, em direção à fita oposta). As nucleobases são combinadas entre os filamentos por meio de ligações de hidrogênio para formar pares de bases. Adenina pareia com timina (duas pontes de hidrogênio) e guanina pareia com citosina (três pontes de hidrogênio).
Fitas de DNA têm uma direcionalidade, e as diferentes extremidades de uma única fita são chamadas de extremidade "3′ (três primos)" e o final "5′ (cinco primos)". Por convenção, se a sequência de bases de uma única fita de DNA for dada, a extremidade esquerda da sequência é a extremidade 5', enquanto a extremidade direita da sequência é a extremidade 3'. As fitas da dupla hélice são antiparalelas, sendo uma de 5' a 3' e a oposta de 3' a 5'. Esses termos referem-se ao átomo de carbono na desoxirribose ao qual o próximo fosfato na cadeia se liga. A direcionalidade tem consequências na síntese de DNA, porque a DNA polimerase pode sintetizar DNA em apenas uma direção adicionando nucleotídeos à extremidade 3' de uma fita de DNA.
O emparelhamento de bases complementares no DNA (através de pontes de hidrogênio) significa que a informação contida em cada fita é redundante. As ligações fosfodiéster (intra-cadeia) são mais fortes do que as ligações de hidrogênio (inter-cadeia). O trabalho real das ligações fosfodiéster é onde os polímeros de DNA conectam o 5' átomo de carbono de um nucleotídeo ao 3' átomo de carbono de outro nucleotídeo, enquanto as pontes de hidrogênio estabilizam as duplas hélices do DNA ao longo do eixo da hélice, mas não na direção do eixo 1. Isso permite que as fitas sejam separadas umas das outras. Os nucleotídeos em uma fita simples podem, portanto, ser usados para reconstruir nucleotídeos em uma fita parceira recentemente sintetizada.
DNA polimerase
As DNA polimerases são uma família de enzimas que realizam todas as formas de replicação do DNA. As DNA polimerases em geral não podem iniciar a síntese de novas fitas, mas apenas estender uma fita de DNA ou RNA existente emparelhada com uma fita molde. Para iniciar a síntese, um pequeno fragmento de RNA, chamado primer, deve ser criado e pareado com a fita molde de DNA.
A DNA polimerase adiciona uma nova fita de DNA estendendo a extremidade 3' de uma cadeia de nucleotídeos existente, adicionando novos nucleotídeos correspondentes à fita molde, um de cada vez, por meio da criação de ligações fosfodiéster. A energia para esse processo de polimerização do DNA vem da hidrólise das ligações de fosfato de alta energia (fosfoanidrido) entre os três fosfatos ligados a cada base não incorporada. As bases livres com seus grupos fosfato ligados são chamadas de nucleotídeos; em particular, as bases com três grupos fosfato ligados são chamadas de trifosfatos de nucleosídeos. Quando um nucleotídeo está sendo adicionado a uma fita de DNA em crescimento, a formação de uma ligação fosfodiéster entre o fosfato proximal do nucleotídeo à cadeia em crescimento é acompanhada pela hidrólise de uma ligação de fosfato de alta energia com liberação dos dois grupos fosfato distais como um pirofosfato. A hidrólise enzimática do pirofosfato resultante em fosfato inorgânico consome uma segunda ligação de fosfato de alta energia e torna a reação efetivamente irreversível.
Em geral, as DNA polimerases são altamente precisas, com uma taxa de erro intrínseco de menos de um erro para cada 107 nucleotídeos adicionados. Além disso, algumas DNA polimerases também possuem capacidade de revisão; eles podem remover nucleotídeos do final de uma fita crescente para corrigir bases incompatíveis. Finalmente, os mecanismos de reparo de incompatibilidade pós-replicação monitoram o DNA em busca de erros, sendo capazes de distinguir incompatibilidades na fita de DNA recém-sintetizada da sequência da fita original. Juntas, essas três etapas de discriminação permitem fidelidade de replicação de menos de um erro para cada 109 nucleotídeos adicionados.
A taxa de replicação do DNA em uma célula viva foi medida pela primeira vez como a taxa de alongamento do DNA do fago T4 em E infectada por fagos. coli. Durante o período de aumento exponencial do DNA a 37°C, a taxa foi de 749 nucleotídeos por segundo. A taxa de mutação por par de bases por replicação durante a síntese de DNA do fago T4 é de 1,7 por 108.
Processo de replicação
A replicação do DNA, como todos os processos de polimerização biológica, ocorre em três etapas coordenadas e catalisadas enzimaticamente: iniciação, alongamento e terminação.
Iniciação
Para uma célula se dividir, ela deve primeiro replicar seu DNA. A replicação do DNA é um processo de tudo ou nada; assim que a replicação começa, ela prossegue até a conclusão. Uma vez concluída a replicação, ela não ocorre novamente no mesmo ciclo celular. Isso é possível pela divisão de iniciação do complexo de pré-replicação.
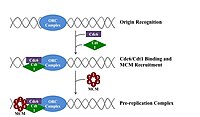
Complexo de pré-replicação
No final da mitose e início da fase G1, um grande complexo de proteínas iniciadoras se reúne no complexo de pré-replicação em pontos específicos do DNA, conhecidos como "origens". Em E. coli a proteína iniciadora primária é DnaA; na levedura, este é o complexo de reconhecimento de origem. As sequências usadas pelas proteínas iniciadoras tendem a ser "ricas em AT" (rico em bases de adenina e timina), porque os pares de bases A-T têm duas ligações de hidrogênio (em vez das três formadas em um par C-G) e, portanto, são mais fáceis de separar em fita. Em eucariotos, o complexo de reconhecimento de origem catalisa a montagem de proteínas iniciadoras no complexo de pré-replicação. Além disso, um relatório recente sugere que a levedura ORC dimeriza de maneira dependente do ciclo celular para controlar o licenciamento. Por sua vez, o processo de dimerização de ORC é mediado por um ciclo de dimerização de Noc3p dependente do ciclo celular in vivo, e esse papel de Noc3p é separável de seu papel na biogênese de ribossomos. [1] ORC e Noc3p estão continuamente ligados à cromatina ao longo do ciclo celular. Cdc6 e Cdt1 então se associam ao complexo de reconhecimento de origem ligado na origem para formar um complexo maior necessário para carregar o complexo Mcm no DNA. O complexo Mcm é a helicase que irá desvendar a hélice de DNA nas origens de replicação e forquilhas de replicação em eucariotos. O complexo Mcm é recrutado no final da fase G1 e carregado pelo complexo ORC-Cdc6-Cdt1 no DNA via remodelação de proteína dependente de ATP. O carregamento do complexo Mcm no DNA de origem marca a conclusão da formação do complexo de pré-replicação.
Se as condições ambientais estiverem corretas no final da fase G1, os complexos G1 e G1/S ciclina-Cdk são ativados, o que estimula a expressão de genes que codificam componentes da maquinaria sintética do DNA. A ativação de G1/S-Cdk também promove a expressão e ativação de complexos S-Cdk, que podem desempenhar um papel na ativação de origens de replicação, dependendo da espécie e do tipo celular. O controle dessas Cdks varia dependendo do tipo de célula e do estágio de desenvolvimento. Esta regulação é melhor compreendida na levedura em brotamento, onde as S ciclinas Clb5 e Clb6 são as principais responsáveis pela replicação do DNA. Os complexos Clb5,6-Cdk1 desencadeiam diretamente a ativação das origens de replicação e, portanto, são necessários ao longo da fase S para ativar diretamente cada origem.
De maneira semelhante, o Cdc7 também é necessário na fase S para ativar as origens de replicação. O Cdc7 não está ativo durante todo o ciclo celular e sua ativação é estritamente cronometrada para evitar o início prematuro da replicação do DNA. No final de G1, a atividade de Cdc7 aumenta abruptamente como resultado da associação com a subunidade reguladora DBF4, que se liga diretamente a Cdc7 e promove sua atividade de proteína quinase. Verificou-se que o Cdc7 é um regulador limitador da taxa da atividade de origem. Juntos, G1/S-Cdks e/ou S-Cdks e Cdc7 colaboram para ativar diretamente as origens de replicação, levando ao início da síntese de DNA.
Complexo de pré-iniciação
No início da fase S, a ativação de S-Cdk e Cdc7 leva à montagem do complexo de pré-iniciação, um complexo proteico maciço formado na origem. A formação do complexo de pré-iniciação desloca Cdc6 e Cdt1 do complexo de replicação de origem, inativando e desmontando o complexo de pré-replicação. Carregar o complexo de pré-iniciação na origem ativa a helicase Mcm, causando o desenrolamento da hélice do DNA. O complexo de pré-iniciação também carrega α-primase e outras DNA polimerases no DNA.
Depois que a α-primase sintetiza os primeiros primers, as junções primer-molde interagem com o clamp loader, que carrega o clamp deslizante no DNA para iniciar a síntese de DNA. Os componentes do complexo de pré-iniciação permanecem associados às forquilhas de replicação à medida que se afastam da origem.
Alongamento
A DNA polimerase tem atividade 5'-3'. Todos os sistemas de replicação de DNA conhecidos requerem um grupo hidroxila 3' livre antes que a síntese possa ser iniciada (nota: o molde de DNA é lido na direção 3' para 5', enquanto uma nova cadeia é sintetizada na direção 5' para 3' - isso geralmente é confuso). Quatro mecanismos distintos para a síntese de DNA são reconhecidos:
- Todas as formas de vida celular e muitos vírus de DNA, fagos e plasmídeos usam uma primase para sintetizar um primer RNA curto com um grupo OH 3′ livre que é posteriormente alongado por uma polimerase de DNA.
- Os retroelementos (incluindo retrovírus) empregam um RNA de transferência que inicia a replicação de DNA fornecendo um OH 3′ livre que é usado para alongamento pela transcriptase reversa.
- Nos adenovírus e na família φ29 de bacteriofages, o grupo 3′ OH é fornecido pela cadeia lateral de um aminoácido do genoma ligado proteína (a proteína terminal) a que nucleotídeos são adicionados pela polimerase do DNA para formar uma nova vertente.
- Nos únicos vírus de DNA encalhados - um grupo que inclui os circovírus, os genminivírus, os parvovírus e outros - e também os muitos fagos e plasmídeos que usam o mecanismo de replicação (RCR), a endonuclease RCR cria um nick na vertente do genoma (vírus isolados) ou uma das vertentes do DNA (plasmídeos). A extremidade 5′ do fio nicked é transferida para um resíduo da tirosina na nuclease e o grupo OH 3′ livre é então usado pela polimerase do DNA para sintetizar o novo fio.
O primeiro é o mais conhecido desses mecanismos e é usado pelos organismos celulares. Nesse mecanismo, uma vez que as duas fitas são separadas, a primase adiciona primers de RNA às fitas-molde. A fita líder recebe um primer de RNA, enquanto a fita atrasada recebe vários. A fita líder é continuamente estendida a partir do primer por uma DNA polimerase com alta processabilidade, enquanto a fita atrasada é estendida descontinuamente a partir de cada primer formando fragmentos de Okazaki. A RNase remove os fragmentos de RNA do iniciador, e uma DNA polimerase de baixa processabilidade distinta da polimerase replicativa entra para preencher as lacunas. Quando isso estiver completo, um único entalhe na fita principal e vários entalhes na fita atrasada podem ser encontrados. A ligase trabalha para preencher esses cortes, completando assim a molécula de DNA recém-replicada.
A primase usada neste processo difere significativamente entre bactérias e archaea/eucariotos. As bactérias usam uma primase pertencente à superfamília de proteínas DnaG que contém um domínio catalítico do tipo TOPRIM fold. A dobra TOPRIM contém um núcleo α/β com quatro fitas conservadas em uma topologia tipo Rossmann. Essa estrutura também é encontrada nos domínios catalíticos da topoisomerase Ia, da topoisomerase II, das nucleases da família OLD e das proteínas de reparo de DNA relacionadas à proteína RecR.
A primase usada por archaea e eucariotos, em contraste, contém uma versão altamente derivada do motivo de reconhecimento de RNA (RRM). Esta primase é estruturalmente semelhante a muitas polimerases de ARN dependentes de ARN virais, transcriptases reversas, ciclases geradoras de nucleótidos cíclicos e polimerases de ADN das famílias A/B/Y que estão envolvidas na replicação e reparação do ADN. Na replicação eucariótica, a primase forma um complexo com Pol α.
Múltiplas DNA polimerases assumem papéis diferentes no processo de replicação do DNA. Em E. coli, DNA Pol III é a enzima polimerase principalmente responsável pela replicação do DNA. Ele se monta em um complexo de replicação na forquilha de replicação que exibe uma processabilidade extremamente alta, permanecendo intacto durante todo o ciclo de replicação. Em contraste, o DNA Pol I é a enzima responsável pela substituição de primers de RNA por DNA. O DNA Pol I tem uma atividade de exonuclease 5' a 3' além de sua atividade de polimerase e usa sua atividade de exonuclease para degradar os iniciadores de RNA à sua frente à medida que estende a fita de DNA atrás dela, em um processo chamado tradução de corte. A Pol I é muito menos processiva do que a Pol III porque sua função primária na replicação do DNA é criar muitas regiões curtas de DNA, em vez de algumas regiões muito longas.
Em eucariotos, a enzima de baixa processabilidade, Pol α, ajuda a iniciar a replicação porque forma um complexo com a primase. Em eucariotos, acredita-se que a síntese do filamento principal seja conduzida por Pol ε; no entanto, essa visão foi recentemente contestada, sugerindo um papel para Pol δ. A remoção do primer é concluída Pol δ enquanto o reparo do DNA durante a replicação é concluído por Pol ε.
À medida que a síntese de DNA continua, os filamentos de DNA originais continuam a se desenrolar em cada lado da bolha, formando uma forquilha de replicação com duas pontas. Nas bactérias, que têm uma única origem de replicação em seu cromossomo circular, esse processo cria uma "estrutura teta" (semelhante à letra grega theta: θ). Em contraste, os eucariotos têm cromossomos lineares mais longos e iniciam a replicação em múltiplas origens dentro deles.
Forquilha de replicação
A forquilha de replicação é uma estrutura que se forma dentro do longo DNA helicoidal durante a replicação do DNA. É criado por helicases, que quebram as pontes de hidrogênio que mantêm as duas fitas de DNA juntas na hélice. A estrutura resultante tem duas "pontas" ramificadas, cada uma composta por uma única fita de DNA. Essas duas fitas servem como modelo para as fitas líder e atrasada, que serão criadas à medida que a DNA polimerase combina os nucleotídeos complementares aos modelos; os moldes podem ser referidos apropriadamente como molde da fita líder e molde da fita atrasada.
O DNA é lido pela DNA polimerase na direção 3' para 5', o que significa que a nova fita é sintetizada na direção 5' para 3' direção. Uma vez que os modelos de fita líder e atrasada são orientados em direções opostas na forquilha de replicação, uma questão importante é como alcançar a síntese de uma nova fita de DNA atrasada, cuja direção de síntese é oposta à direção da replicação crescente garfo.
Vertente principal
A fita líder é a fita do novo DNA que é sintetizado na mesma direção da forquilha de replicação em crescimento. Esse tipo de replicação do DNA é contínuo.
Vertente atrasada
A fita retardada é a fita do novo DNA cuja direção de síntese é oposta à direção da forquilha de replicação em crescimento. Devido à sua orientação, a replicação da fita retardada é mais complicada em comparação com a da fita líder. Como consequência, a DNA polimerase nesta fita parece "ficar para trás" a outra mecha.
A fita retardada é sintetizada em segmentos curtos e separados. No template da vertente atrasada, uma primase "lê" o DNA molde e inicia a síntese de um pequeno iniciador de RNA complementar. Uma DNA polimerase estende os segmentos iniciados, formando fragmentos de Okazaki. Os primers de RNA são então removidos e substituídos por DNA, e os fragmentos de DNA são unidos pela DNA ligase.
Dinâmica na forquilha de replicação
Em todos os casos, a helicase é composta por seis polipeptídeos que envolvem apenas uma fita do DNA que está sendo replicado. As duas polimerases estão ligadas ao heximero da helicase. Nos eucariotos, a helicase envolve a fita líder e, nos procariontes, ela envolve a fita atrasada.
Conforme a helicase desenrola o DNA na forquilha de replicação, o DNA à frente é forçado a girar. Esse processo resulta em um acúmulo de reviravoltas no DNA à frente. Esse acúmulo forma uma resistência à torção que acabaria por interromper o progresso da forquilha de replicação. As topoisomerases são enzimas que quebram temporariamente as fitas de DNA, aliviando a tensão causada pelo desenrolamento das duas fitas da hélice de DNA; as topoisomerases (incluindo a DNA girase) conseguem isso adicionando superenrolamentos negativos à hélice do DNA.
O DNA de cadeia simples nua tende a dobrar-se sobre si mesmo, formando estruturas secundárias; essas estruturas podem interferir no movimento da DNA polimerase. Para evitar isso, proteínas de ligação de fita simples se ligam ao DNA até que uma segunda fita seja sintetizada, evitando a formação de estruturas secundárias.
O DNA de fita dupla é enrolado em torno de histonas que desempenham um papel importante na regulação da expressão gênica, de modo que o DNA replicado deve ser enrolado em torno de histonas nos mesmos lugares que o DNA original. Para garantir isso, as acompanhantes de histonas desmontam a cromatina antes que ela seja replicada e recolocam as histonas no lugar correto. Algumas etapas nesta remontagem são um tanto especulativas.
As proteínas de fixação formam uma fixação deslizante em torno do DNA, ajudando a DNA polimerase a manter contato com seu modelo, auxiliando assim na processabilidade. A face interna da braçadeira permite que o DNA seja passado por ela. Uma vez que a polimerase atinge o final do molde ou detecta DNA de fita dupla, o grampo deslizante sofre uma mudança conformacional que libera a DNA polimerase. As proteínas de carregamento de grampo são usadas para carregar inicialmente o grampo, reconhecendo a junção entre o modelo e os primers de RNA.:274-5
Proteínas de replicação de DNA
Na forquilha de replicação, muitas enzimas de replicação se reúnem no DNA em uma máquina molecular complexa chamada replissoma. A seguir está uma lista das principais enzimas de replicação do DNA que participam do replissoma:
| Enzima | Função na replicação de DNA |
|---|---|
| Helicase de DNA | Também conhecido como enzima desestabilizadora de hélice. A helicase separa as duas vertentes do DNA no Fork de Replicação atrás da topoisomerase. |
| Polimerase de DNA | A enzima responsável por catalisar a adição de substratos nucleotídeos ao DNA na direção 5′ a 3′ durante a replicação do DNA. Também executa a correção de leitura e erro. Existem muitos tipos diferentes de polimerase de DNA, cada um deles executa funções diferentes em diferentes tipos de células. |
| Grampo de ADN | Uma proteína que impede o alongamento do DNA polimerases de dissociar a partir da cadeia pai do DNA. |
| Proteína de ligação de DNA única | Ligado ao ssDNA e evitar que a dupla hélice do DNA re-recolhe após a helicase do DNA desbobiná-lo, mantendo assim a separação da cadeia, e facilitando a síntese da nova cadeia. |
| Topoisomerase | Relaxa o ADN da sua natureza super cozida. |
| Girase de DNA | Alivia a tensão de desenrolar por helicase de DNA; este é um tipo específico de topoisomerase |
| Ligação do ADN | Recolhe os fios semi-conservadores e junta-se aos Fragmentos Okazaki do fio de lagging. |
| Primaz | Fornece um ponto de partida de RNA (ou DNA) para a polimerase de DNA para começar a síntese da nova cadeia de DNA. |
| Linha de produção | Alonga o DNA telomérico adicionando sequências de nucleotídeos repetitivas às extremidades de cromossomas eucarióticos. Isso permite que as células germinativas e células-tronco evitem o limite de Hayflick na divisão celular. |
Máquina de replicação
Máquinas de replicação consistem em fatores envolvidos na replicação do DNA e que aparecem no modelo ssDNAs. Máquinas de replicação incluem primosotors são enzimas de replicação; DNA polimerase, DNA helicases, DNA clamps e DNA topoisomerases e proteínas de replicação; por exemplo. proteínas de ligação ao DNA de cadeia simples (SSB). Nas máquinas de replicação, esses componentes se coordenam. Na maioria das bactérias, todos os fatores envolvidos na replicação do DNA estão localizados nas forquilhas de replicação e os complexos permanecem nas forquilhas durante a replicação do DNA. Esses mecanismos de replicação são chamados de replissomos ou sistemas de replicação de DNA. Esses termos são termos genéricos para proteínas localizadas nas forquilhas de replicação. Em células eucarióticas e algumas bacterianas, os replissomas não são formados.
Como as máquinas de replicação não se movem relativamente aos DNAs modelo, como fábricas, elas são chamadas de fábrica de replicação. Em uma figura alternativa, as fábricas de DNA são semelhantes a projetores e os DNAs são como filmes cinematográficos passando constantemente nos projetores. No modelo da fábrica de replicação, depois que ambas as helicases de DNA para fitas principais e fitas atrasadas são carregadas nos DNAs modelo, as helicases correm ao longo dos DNAs umas nas outras. As helicases permanecem associadas pelo restante do processo de replicação. Peter Meister e outros. observaram diretamente os locais de replicação em leveduras de brotamento monitorando DNA polimerases α marcadas com proteína verde fluorescente (GFP). Eles detectaram a replicação do DNA de pares de loci marcados espaçados simetricamente de uma origem de replicação e descobriram que a distância entre os pares diminuiu acentuadamente com o tempo. Esta descoberta sugere que o mecanismo de replicação do DNA acompanha as fábricas de DNA. Ou seja, pares de fábricas de replicação são carregados nas origens de replicação e nas fábricas associadas entre si. Além disso, os DNAs modelo se movem para as fábricas, que trazem a extrusão dos ssDNAs modelo e novos DNAs. A descoberta de Meister é a primeira evidência direta do modelo de fábrica de replicação. Pesquisas subsequentes mostraram que as helicases de DNA formam dímeros em muitas células eucarióticas e as máquinas de replicação bacteriana permanecem em um único local intranuclear durante a síntese de DNA.
As fábricas de replicação realizam o desemaranhamento das cromátides-irmãs. O desemaranhamento é essencial para a distribuição das cromátides nas células-filhas após a replicação do DNA. Como as cromátides irmãs após a replicação do DNA se mantêm por anéis de coesina, existe a única chance de desemaranhamento na replicação do DNA. A fixação de máquinas de replicação como fábricas de replicação pode melhorar a taxa de sucesso da replicação do DNA. Se as forquilhas de replicação se moverem livremente nos cromossomos, a catenação dos núcleos é agravada e impede a segregação mitótica.
Rescisão
Os eucariotos iniciam a replicação do DNA em vários pontos do cromossomo, de modo que os garfos de replicação se encontram e terminam em muitos pontos do cromossomo. Como os eucariotos têm cromossomos lineares, a replicação do DNA é incapaz de atingir o final dos cromossomos. Devido a esse problema, o DNA é perdido em cada ciclo de replicação a partir do final do cromossomo. Os telômeros são regiões de DNA repetitivo próximas às pontas e ajudam a evitar a perda de genes devido a esse encurtamento. O encurtamento dos telômeros é um processo normal nas células somáticas. Isso encurta os telômeros do cromossomo filho do DNA. Como resultado, as células só podem se dividir um certo número de vezes antes que a perda de DNA impeça a divisão. (Isso é conhecido como limite de Hayflick.) Dentro da linhagem de células germinativas, que passa o DNA para a próxima geração, a telomerase estende as sequências repetitivas da região dos telômeros para evitar a degradação. A telomerase pode se tornar erroneamente ativa nas células somáticas, às vezes levando à formação de câncer. O aumento da atividade da telomerase é uma das características do câncer.
O término requer que o progresso da forquilha de replicação do DNA seja interrompido ou bloqueado. A terminação em um locus específico, quando ocorre, envolve a interação entre dois componentes: (1) uma sequência de local de terminação no DNA e (2) uma proteína que se liga a essa sequência para interromper fisicamente a replicação do DNA. Em várias espécies bacterianas, isso é chamado de proteína de ligação ao local do terminal de replicação do DNA, ou proteína Ter.
Como as bactérias têm cromossomos circulares, o término da replicação ocorre quando as duas forquilhas de replicação se encontram na extremidade oposta do cromossomo parental. E. coli regula esse processo por meio do uso de sequências de terminação que, quando ligadas pela proteína Tus, permitem a passagem de apenas uma direção da forquilha de replicação. Como resultado, as forquilhas de replicação são obrigadas a sempre se encontrarem na região de terminação do cromossomo.
Regulamento
Eucarióticas
Nos eucariotos, a replicação do DNA é controlada dentro do contexto do ciclo celular. À medida que a célula cresce e se divide, ela progride através dos estágios do ciclo celular; A replicação do DNA ocorre durante a fase S (fase de síntese). O progresso da célula eucariótica através do ciclo é controlado por pontos de verificação do ciclo celular. A progressão através dos checkpoints é controlada por meio de interações complexas entre várias proteínas, incluindo ciclinas e quinases dependentes de ciclina. Ao contrário das bactérias, o DNA eucariótico se replica nos confins do núcleo.
O ponto de verificação G1/S (ou ponto de verificação de restrição) regula se as células eucarióticas entram no processo de replicação do DNA e subseqüente divisão. As células que não passam por esse ponto de verificação permanecem no estágio G0 e não replicam seu DNA.
Depois de passar pelo ponto de verificação G1/S, o DNA deve ser replicado apenas uma vez em cada ciclo celular. Quando o complexo Mcm se afasta da origem, o complexo de pré-replicação é desmantelado. Como um novo complexo Mcm não pode ser carregado em uma origem até que as subunidades de pré-replicação sejam reativadas, uma origem de replicação não pode ser usada duas vezes no mesmo ciclo celular.
A ativação de S-Cdks no início da fase S promove a destruição ou inibição de componentes individuais do complexo de pré-replicação, impedindo a remontagem imediata. S e M-Cdks continuam a bloquear a montagem do complexo de pré-replicação mesmo após a conclusão da fase S, garantindo que a montagem não ocorra novamente até que toda a atividade de Cdk seja reduzida na mitose tardia.
Na levedura de brotamento, a inibição da montagem é causada pela fosforilação dependente de Cdk de componentes do complexo de pré-replicação. No início da fase S, a fosforilação de Cdc6 por Cdk1 causa a ligação de Cdc6 à proteína ligase SCF ubiquitina, que causa a destruição proteolítica de Cdc6. A fosforilação dependente de Cdk de proteínas Mcm promove sua exportação para fora do núcleo junto com Cdt1 durante a fase S, impedindo o carregamento de novos complexos Mcm nas origens durante um único ciclo celular. A fosforilação de Cdk do complexo de replicação de origem também inibe a montagem do complexo de pré-replicação. A presença individual de qualquer um desses três mecanismos é suficiente para inibir a montagem do complexo pré-replicação. No entanto, as mutações de todas as três proteínas na mesma célula desencadeiam a reinicialização em muitas origens de replicação dentro de um ciclo celular.
Em células animais, a proteína geminina é um inibidor chave da montagem do complexo de pré-replicação. Geminina se liga a Cdt1, impedindo sua ligação ao complexo de reconhecimento de origem. Em G1, os níveis de geminina são mantidos baixos pelo APC, que ubiquitina a geminina para direcioná-la para a degradação. Quando o geminin é destruído, o Cdt1 é liberado, permitindo que ele funcione na montagem complexa de pré-replicação. No final de G1, o APC é inativado, permitindo que a geminina se acumule e se ligue a Cdt1.
A replicação dos genomas dos cloroplastos e mitocondriais ocorre independentemente do ciclo celular, através do processo de replicação D-loop.
Foco na replicação
Em células de vertebrados, os locais de replicação se concentram em posições chamadas focos de replicação. Os locais de replicação podem ser detectados por cadeias filhas de imunocoloração e enzimas de replicação e monitoramento de fatores de replicação marcados com GFP. Por esses métodos, descobriu-se que os focos de replicação de tamanhos e posições variáveis aparecem na fase S da divisão celular e seu número por núcleo é muito menor do que o número de forquilhas de replicação genômica.
P. Heun et al., (2001) rastreou focos de replicação marcados com GFP em células de levedura em brotamento e revelou que as origens de replicação se movem constantemente nas fases G1 e S e a dinâmica diminuiu significativamente na fase S. Tradicionalmente, os sítios de replicação eram fixados na estrutura espacial dos cromossomos pela matriz nuclear ou lâminas. Os resultados de Heun refutam os conceitos tradicionais, leveduras em brotamento não possuem laminas, e sustentam que as origens de replicação se auto-montam e formam focos de replicação.
Ao disparar as origens de replicação, controladas espacial e temporalmente, a formação de focos de replicação é regulada. D. A. Jackson et al. (1998) revelou que origens vizinhas disparam simultaneamente em células de mamíferos. A justaposição espacial de locais de replicação traz agrupamento de forquilhas de replicação. O agrupamento resgata as forquilhas de replicação paralisadas e favorece o progresso normal das forquilhas de replicação. O progresso das forquilhas de replicação é inibido por muitos fatores; colisão com proteínas ou com complexos que se ligam fortemente ao DNA, deficiência de dNTPs, cortes nos DNAs modelo e assim por diante. Se as forquilhas de replicação pararem e as sequências restantes das forquilhas paralisadas não forem replicadas, as fitas-filhas cortaram os locais não replicados obtidos. Os locais não replicados na fita de um dos pais mantêm a outra fita unida, mas não as fitas filhas. Portanto, as cromátides-irmãs resultantes não podem se separar umas das outras e não podem se dividir em 2 células-filhas. Quando as origens vizinhas disparam e uma bifurcação de uma origem está parada, a bifurcação de outra origem acessa na direção oposta da bifurcação parada e duplica os sites não replicados. Como outro mecanismo de resgate, existe a aplicação de origens de replicação dormentes para que as origens em excesso não sejam acionadas na replicação normal do DNA.
Bactérias
A maioria das bactérias não passa por um ciclo celular bem definido, mas copia continuamente seu DNA; durante o crescimento rápido, isso pode resultar na ocorrência simultânea de várias rodadas de replicação. Em E. coli, a bactéria mais bem caracterizada, a replicação do DNA é regulada por vários mecanismos, incluindo: a hemimetilação e o sequestro da sequência de origem, a proporção de adenosina trifosfato (ATP) para adenosina difosfato (ADP) e os níveis de proteína DnaA. Todos estes controlam a ligação das proteínas iniciadoras às sequências de origem.
Porque E. coli metila sequências de DNA GATC, a síntese de DNA resulta em sequências hemimetiladas. Esse DNA hemimetilado é reconhecido pela proteína SeqA, que se liga e sequestra a sequência de origem; além disso, o DnaA (necessário para o início da replicação) liga-se menos bem ao DNA hemimetilado. Como resultado, as origens recém-replicadas são impedidas de iniciar imediatamente outra rodada de replicação do DNA.
O ATP se acumula quando a célula está em um meio rico, desencadeando a replicação do DNA assim que a célula atinge um tamanho específico. O ATP compete com o ADP para se ligar ao DnaA, e o complexo DnaA-ATP é capaz de iniciar a replicação. Um certo número de proteínas DnaA também é necessário para a replicação do DNA - cada vez que a origem é copiada, o número de sítios de ligação para o DnaA dobra, exigindo a síntese de mais DnaA para permitir outro início de replicação.
Em bactérias de crescimento rápido, como E. coli, a replicação do cromossomo leva mais tempo do que a divisão da célula. A bactéria resolve isso iniciando uma nova rodada de replicação antes que a anterior tenha terminado. A nova rodada de replicação formará o cromossomo da célula que nasce duas gerações após a célula em divisão. Esse mecanismo cria ciclos de replicação sobrepostos.
Problemas com a replicação do DNA
Existem muitos eventos que contribuem para o estresse de replicação, incluindo:
- Misincorporação de ribonucleotides
- Estruturas de DNA incomuns
- Conflitos entre replicação e transcrição
- Insuficiência de fatores de replicação essenciais
- Locais frágeis comuns
- Expressão ou ativação constitutiva de oncogenes
- Inacessibilidade de cromatina
Reação em cadeia da polimerase
Os pesquisadores geralmente replicam o DNA in vitro usando a reação em cadeia da polimerase (PCR). A PCR usa um par de iniciadores para abranger uma região alvo no DNA modelo e, em seguida, polimeriza as cadeias parceiras em cada direção a partir desses iniciadores usando uma polimerase de DNA termoestável. A repetição desse processo por vários ciclos amplifica a região de DNA alvo. No início de cada ciclo, a mistura de molde e primers é aquecida, separando a molécula recém-sintetizada e o molde. Então, à medida que a mistura esfria, ambos se tornam modelos para recozimento de novos primers, e a polimerase se estende a partir deles. Como resultado, o número de cópias da região-alvo dobra a cada rodada, aumentando exponencialmente.
Contenido relacionado
Quadrupedalismo
Mamífero
Enzima