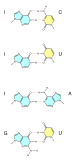
Par base
Um par de bases (bp) é uma unidade fundamental de ácidos nucléicos de fita dupla que consiste em duas nucleobases ligadas entre si por pontes de hidrogênio. Eles formam os blocos de construção da dupla hélice do DNA e contribuem para a estrutura dobrada do DNA e do RNA. Ditado por padrões específicos de ligação de hidrogênio, "Watson-Crick" (ou "Watson-Crick-Franklin") pares de bases (guanina-citosina e adenina-timina) permitem que a hélice de DNA mantenha uma estrutura helicoidal regular que é sutilmente dependente de sua sequência de nucleotídeos. A natureza complementar desta estrutura baseada em pares fornece uma cópia redundante da informação genética codificada em cada fita de DNA. A estrutura regular e a redundância de dados fornecidas pela dupla hélice do DNA tornam o DNA adequado para o armazenamento de informações genéticas, enquanto o pareamento de bases entre o DNA e os nucleotídeos recebidos fornece o mecanismo através do qual a DNA polimerase replica o DNA e a RNA polimerase transcreve o DNA em RNA. Muitas proteínas de ligação ao DNA podem reconhecer padrões específicos de pareamento de bases que identificam regiões reguladoras particulares de genes.
Os pares de bases intramoleculares podem ocorrer em ácidos nucleicos de cadeia simples. Isso é particularmente importante em moléculas de RNA (por exemplo, RNA de transferência), onde os pares de bases Watson-Crick (guanina-citosina e adenina-uracil) permitem a formação de hélices duplas curtas e uma ampla variedade de interações não-Watson-Crick (por exemplo, G-U ou A-A) permitem que os RNAs se dobrem em uma vasta gama de estruturas tridimensionais específicas. Além disso, o pareamento de bases entre o RNA de transferência (tRNA) e o RNA mensageiro (mRNA) forma a base para os eventos de reconhecimento molecular que resultam na tradução da sequência de nucleotídeos do mRNA na sequência de aminoácidos das proteínas por meio do código genético.
O tamanho de um gene individual ou de todo o genoma de um organismo geralmente é medido em pares de bases porque o DNA geralmente é de fita dupla. Portanto, o número total de pares de bases é igual ao número de nucleotídeos em uma das fitas (com exceção das regiões de fita simples não codificantes dos telômeros). Estima-se que o genoma humano haploide (23 cromossomos) tenha cerca de 3,2 bilhões de bases e contenha 20.000 a 25.000 genes codificadores de proteínas distintos. Uma quilobase (kb) é uma unidade de medida em biologia molecular igual a 1000 pares de bases de DNA ou RNA. O número total de pares de bases de DNA na Terra é estimado em 5,0×1037 com um peso de 50 bilhões de toneladas. Em comparação, a massa total da biosfera foi estimada em até 4 TtC (trilhões de toneladas de carbono).
Ligação de hidrogênio e estabilidade
 |
 |
A ligação de hidrogênio é a interação química subjacente às regras de pareamento de bases descritas acima. A correspondência geométrica apropriada de doadores e aceitadores de ligações de hidrogênio permite apenas a correspondência "certa" pares para formar de forma estável. O DNA com alto conteúdo de GC é mais estável do que o DNA com baixo conteúdo de GC. Mas, ao contrário da crença popular, as pontes de hidrogênio não estabilizam significativamente o DNA; estabilização é principalmente devido a interações de empilhamento.
As nucleobases maiores, adenina e guanina, são membros de uma classe de estruturas químicas com anéis duplos chamadas purinas; as nucleobases menores, citosina e timina (e uracil), são membros de uma classe de estruturas químicas de anel único chamadas pirimidinas. As purinas são complementares apenas com as pirimidinas: os pareamentos pirimidina-pirimidina são energeticamente desfavoráveis porque as moléculas estão muito distantes umas das outras para que ligações de hidrogênio sejam estabelecidas; Os pareamentos purina-purina são energeticamente desfavoráveis porque as moléculas estão muito próximas, levando à repulsão por sobreposição. O pareamento de bases purina-pirimidina de AT ou GC ou UA (no RNA) resulta em uma estrutura duplex adequada. Os únicos outros pareamentos purina-pirimidina seriam AC e GT e UG (no RNA); esses pareamentos são incompatíveis porque os padrões de doadores e aceitadores de hidrogênio não correspondem. O emparelhamento GU, com duas ligações de hidrogênio, ocorre com bastante frequência no RNA (consulte par de bases oscilantes).
Moléculas pareadas de DNA e RNA são comparativamente estáveis à temperatura ambiente, mas as duas fitas de nucleotídeos se separarão acima de um ponto de fusão determinado pelo comprimento das moléculas, a extensão do pareamento incorreto (se houver) e o conteúdo de GC. Maior conteúdo de GC resulta em temperaturas de fusão mais altas; não é, portanto, surpreendente que os genomas de organismos extremófilos como Thermus thermophilus sejam particularmente ricos em GC. Por outro lado, as regiões de um genoma que precisam se separar com frequência - por exemplo, as regiões promotoras de genes frequentemente transcritos - são comparativamente pobres em GC (por exemplo, consulte a caixa TATA). O conteúdo de GC e a temperatura de fusão também devem ser levados em consideração ao projetar primers para reações de PCR.
Exemplos
As sequências de DNA a seguir ilustram padrões de pares de fita dupla. Por convenção, a fita superior é escrita da extremidade 5' para a extremidade 3'; assim, a fita inferior é escrita de 3' a 5'.
- Uma sequência de ADN de base:
ATCGATTGAGCTCTAGCGTAGCTAACTCGAGATCGC
- A sequência de RNA correspondente, em que o uracil é substituído por timina no fio do RNA:
AUCGAUUGAGCUCUAGCGUAGCUAACUCGAGAUCGC
Análogos de base e intercaladores
Análogos químicos de nucleotídeos podem substituir nucleotídeos apropriados e estabelecer pareamento de bases não canônico, levando a erros (principalmente mutações pontuais) na replicação e transcrição do DNA. Isto é devido à sua química isostérica. Um análogo comum de base mutagênica é o 5-bromouracil, que se assemelha à timina, mas pode parear com a guanina em sua forma enol.
Outras substâncias químicas, conhecidas como intercaladores de DNA, se encaixam no espaço entre bases adjacentes em uma única fita e induzem mutações frameshift por "mascaramento" como uma base, fazendo com que a maquinaria de replicação do DNA pule ou insira nucleotídeos adicionais no sítio intercalado. A maioria dos intercaladores são grandes compostos poliaromáticos e são cancerígenos conhecidos ou suspeitos. Exemplos incluem brometo de etídio e acridina.
Reparo de incompatibilidade
Pares de bases incompatíveis podem ser gerados por erros de replicação do DNA e como intermediários durante a recombinação homóloga. O processo de reparo de incompatibilidade normalmente deve reconhecer e reparar corretamente um pequeno número de pares de base incorretos dentro de uma longa sequência de pares de bases de DNA normais. Para reparar incompatibilidades formadas durante a replicação do DNA, vários processos distintos de reparo evoluíram para distinguir entre a fita molde e a fita recém-formada, de modo que apenas o nucleotídeo incorreto inserido recentemente seja removido (para evitar a geração de uma mutação). As proteínas empregadas no reparo de incompatibilidade durante a replicação do DNA e o significado clínico dos defeitos nesse processo são descritos no artigo reparo de incompatibilidade de DNA. O processo de correção de pares incorretos durante a recombinação é descrito no artigo conversão de genes.
Medições de comprimento
As seguintes abreviações são comumente usadas para descrever o comprimento de uma molécula de D/RNA:
- bp = par base—um bp corresponde a aproximadamente 3,4 Å (340 pm) de comprimento ao longo da cadeia, e a aproximadamente 618 ou 643 daltons para DNA e RNA respectivamente.
- kb (= kbp) = quilo-base-pair = 1.000 bp
- Mb (= Mbp) = mega-base-pair = 1.000.000 bp
- Gb = giga-base-pair = 1.000.000,000 bp.
Para DNA/RNA de fita simples, são usadas unidades de nucleotídeos—abreviados nt (ou knt, Mnt, Gnt)—pois eles não são pareados. Para distinguir entre unidades de armazenamento de computador e bases, kbp, Mbp, Gbp, etc. podem ser usados para pares de bases.
O centimorgan também é frequentemente usado para indicar a distância ao longo de um cromossomo, mas o número de pares de bases aos quais ele corresponde varia muito. No genoma humano, o centimorgan tem cerca de 1 milhão de pares de bases.
Par de base não natural (UBP)
Um par de bases não naturais (UBP) é uma subunidade projetada (ou nucleobase) de DNA que é criada em laboratório e não ocorre na natureza. Foram descritas sequências de DNA que utilizam nucleobases recém-criadas para formar um terceiro par de bases, além dos dois pares de bases encontrados na natureza, A-T (adenina – timina) e G-C (guanina – citosina). Alguns grupos de pesquisa têm procurado um terceiro par de bases para o DNA, incluindo equipes lideradas por Steven A. Benner, Philippe Marliere, Floyd E. Romesberg e Ichiro Hirao. Alguns novos pares de bases baseados em ligações de hidrogênio alternativas, interações hidrofóbicas e coordenação de metais foram relatados.
Em 1989, Steven Benner (então trabalhando no Instituto Federal Suíço de Tecnologia em Zurique) e sua equipe lideraram com formas modificadas de citosina e guanina em moléculas de DNA in vitro. Os nucleotídeos, que codificam RNA e proteínas, foram replicados com sucesso in vitro. Desde então, a equipe de Benner tem tentado projetar células que possam fazer bases estrangeiras a partir do zero, eliminando a necessidade de uma matéria-prima.
Em 2002, o grupo de Ichiro Hirao no Japão desenvolveu um par de bases não naturais entre 2-amino-8-(2-tienil)purina (s) e piridina-2-ona (y) que funciona na transcrição e tradução, para a incorporação site-specific de aminoácidos não padronizados em proteínas. Em 2006, eles criaram 7-(2-tienil)imidazo[4,5-b]piridina (Ds) e pirrole-2-carbaldeído (Pa) como um terceiro par de bases para replicação e transcrição. Posteriormente, Ds e 4-[3-(6-aminohexanamido)-1-propinil]-2-nitropirrol (Px) foram descobertos como um par de alta fidelidade na amplificação por PCR. Em 2013, eles aplicaram o par Ds-Px à geração de aptâmeros de DNA por seleção in vitro (SELEX) e demonstraram que a expansão do alfabeto genético aumenta significativamente as afinidades dos aptâmeros de DNA com as proteínas-alvo.
Em 2012, um grupo de cientistas americanos liderados por Floyd Romesberg, um biólogo químico do Scripps Research Institute em San Diego, Califórnia, publicou que sua equipe projetou um par de bases não naturais (UBP). Os dois novos nucleotídeos artificiais ou Unnatural Base Pair (UBP) foram denominados d5SICS e dNaM. Mais tecnicamente, esses nucleotídeos artificiais contendo nucleobases hidrofóbicas apresentam dois anéis aromáticos fundidos que formam um complexo (d5SICS-dNaM) ou par de bases no DNA. Sua equipe projetou uma variedade de testes in vitro ou "tubo de ensaio" modelos contendo o par de bases não naturais e eles confirmaram que ele foi replicado de forma eficiente com alta fidelidade em praticamente todos os contextos de sequência usando as técnicas modernas in vitro padrão, ou seja, amplificação de PCR de DNA e aplicações baseadas em PCR. Seus resultados mostram que para PCR e aplicações baseadas em PCR, o par de bases não naturais d5SICS-dNaM é funcionalmente equivalente a um par de bases naturais, e quando combinado com os outros dois pares de bases naturais usados por todos os organismos, A-T e G-C, eles fornecem um "alfabeto genético" de seis letras totalmente funcional e expandido.
Em 2014, a mesma equipe do Scripps Research Institute relatou que sintetizou um trecho de DNA circular conhecido como plasmídeo contendo pares de bases T-A e C-G naturais junto com o laboratório de UBP Romesberg de melhor desempenho projetado e inserido em células da bactéria comum E. coli que replicou com sucesso os pares de bases não naturais através de múltiplas gerações. A transfecção não prejudicou o crescimento de E. coli e não mostrou nenhum sinal de perda de seus pares de bases não naturais para seus mecanismos naturais de reparo de DNA. Este é o primeiro exemplo conhecido de um organismo vivo transmitindo um código genético expandido para as gerações subsequentes. Romesberg disse que ele e seus colegas criaram 300 variantes para refinar o design de nucleotídeos que seriam estáveis o suficiente e seriam replicados tão facilmente quanto os naturais quando as células se dividissem. Isto foi em parte alcançado pela adição de um gene de alga de suporte que expressa um transportador de trifosfato de nucleotídeo que importa eficientemente os trifosfatos de d5SICSTP e dNaMTP para E. coli bactérias. Em seguida, as vias naturais de replicação bacteriana os usam para replicar com precisão um plasmídeo contendo d5SICS-dNaM. Outros pesquisadores ficaram surpresos com o fato de a bactéria replicar essas subunidades de DNA feitas pelo homem.
A incorporação bem-sucedida de um terceiro par de bases é um avanço significativo em direção ao objetivo de expandir consideravelmente o número de aminoácidos que podem ser codificados pelo DNA, dos 20 aminoácidos existentes para teoricamente possíveis 172, expandindo assim o potencial para organismos vivos para produzir novas proteínas. As cadeias artificiais de DNA ainda não codificam nada, mas os cientistas especulam que elas podem ser projetadas para fabricar novas proteínas que podem ter usos industriais ou farmacêuticos. Especialistas disseram que o DNA sintético que incorpora o par de bases não naturais levanta a possibilidade de formas de vida baseadas em um código de DNA diferente.
Emparelhamento de base não canônico
Além do emparelhamento canônico, algumas condições também podem favorecer o emparelhamento de bases com orientação de base alternativa e número e geometria de ligações de hidrogênio. Esses pareamentos são acompanhados por alterações na forma da espinha dorsal local.
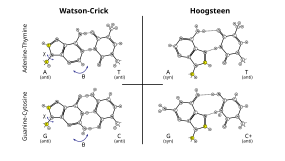
O mais comum deles é o pareamento oscilante de bases que ocorre entre tRNAs e mRNAs na terceira posição de base de muitos códons durante a transcrição e durante o carregamento de tRNAs por algumas sintetases de tRNA. Eles também foram observados nas estruturas secundárias de algumas sequências de RNA.
Além disso, o pareamento de bases Hoogsteen (normalmente escrito como A•U/T e G•C) pode existir em algumas sequências de DNA (por exemplo, dinucleotídeos CA e TA) em equilíbrio dinâmico com o pareamento Watson–Crick padrão. Eles também foram observados em alguns complexos proteína-DNA.
Além desses pares alternativos de bases, uma ampla gama de pontes de hidrogênio base-base é observada na estrutura secundária e terciária do RNA. Essas ligações são muitas vezes necessárias para a forma precisa e complexa de um RNA, bem como sua ligação aos parceiros de interação.
Contenido relacionado
Amigdalina
Amarílis
Arecales