Fator de transcrição

Em biologia molecular, um fator de transcrição (TF) (ou fator de ligação ao DNA específico da sequência) é uma proteína que controla o taxa de transcrição da informação genética do DNA para o RNA mensageiro, por ligação a uma sequência específica de DNA. A função dos TFs é regular – ligar e desligar – genes para garantir que eles sejam expressos nas células desejadas, no momento certo e na quantidade certa ao longo da vida da célula e do organismo. Grupos de TFs funcionam de forma coordenada para direcionar a divisão celular, o crescimento celular e a morte celular ao longo da vida; migração e organização celular (plano corporal) durante o desenvolvimento embrionário; e intermitentemente em resposta a sinais externos à célula, como um hormônio. Existem 1.500-1.600 TFs no genoma humano. Os fatores de transcrição são membros do proteoma e também do reguloma.
Os TFs funcionam sozinhos ou com outras proteínas em um complexo, promovendo (como um ativador) ou bloqueando (como um repressor) o recrutamento da RNA polimerase (a enzima que realiza a transcrição da informação genética do DNA para o RNA) para genes específicos.
Uma característica definidora dos TFs é que eles contêm pelo menos um domínio de ligação ao DNA (DBD), que se liga a uma sequência específica de DNA adjacente aos genes que eles regulam. Os TFs são agrupados em classes com base em seus DBDs. Outras proteínas, como coativadores, remodeladores de cromatina, histonas acetiltransferases, histonas desacetilases, quinases e metilases também são essenciais para a regulação gênica, mas não possuem domínios de ligação ao DNA e, portanto, não são TFs.
Os TFs são de interesse na medicina porque as mutações dos TFs podem causar doenças específicas e os medicamentos podem ser potencialmente direcionados a elas.
Número
Os factores de transcrição são essenciais para a regulação da expressão genética e são, como consequência, encontrados em todos os organismos vivos. O número de fatores de transcrição encontrados em um organismo aumenta com o tamanho do genoma, e genomas maiores tendem a ter mais fatores de transcrição por gene.
Existem aproximadamente 2.800 proteínas no genoma humano que contêm domínios de ligação ao DNA, e presume-se que 1.600 delas funcionem como fatores de transcrição, embora outros estudos indiquem que seja um número menor. Portanto, aproximadamente 10% dos genes no genoma codificam fatores de transcrição, o que torna esta família a maior família de proteínas humanas. Além disso, os genes são frequentemente flanqueados por vários locais de ligação para factores de transcrição distintos, e a expressão eficiente de cada um destes genes requer a acção cooperativa de vários factores de transcrição diferentes (ver, por exemplo, factores nucleares de hepatócitos). Assim, o uso combinatório de um subconjunto de aproximadamente 2.000 fatores de transcrição humanos é facilmente responsável pela regulação única de cada gene no genoma humano durante o desenvolvimento.
Mecanismo
Os fatores de transcrição ligam-se às regiões intensificadoras ou promotoras do DNA adjacentes aos genes que eles regulam. Dependendo do fator de transcrição, a transcrição do gene adjacente é regulada para cima ou para baixo. Os fatores de transcrição utilizam uma variedade de mecanismos para a regulação da expressão gênica. Esses mecanismos incluem:
- estabilizar ou bloquear a ligação da polimerase do RNA ao ADN
- catalisar a acetilação ou deacetilação de proteínas histone. O fator de transcrição pode fazer isso diretamente ou recrutar outras proteínas com esta atividade catalítica. Muitos fatores de transcrição usam um ou outro de dois mecanismos opostos para regular a transcrição:
- atividade de acetiltransferase de histone (HAT) - acetilatos proteínas histone, que enfraquece a associação de DNA com histones, o que torna o DNA mais acessível à transcrição, assim transcrição up-regulating
- histone deacetilase (HDAC) atividade - deacetila as proteínas histone, que fortalece a associação de DNA com histones, o que torna o DNA menos acessível à transcrição, assim, transcrição de regulação para baixo
- recrutar proteína coativadora ou corepressor para o complexo de DNA do fator de transcrição
Função
Os fatores de transcrição são um dos grupos de proteínas que leem e interpretam o "modelo" genético. no ADN. Eles se ligam ao DNA e ajudam a iniciar um programa de aumento ou diminuição da transcrição genética. Como tal, são vitais para muitos processos celulares importantes. Abaixo estão algumas das funções importantes e papéis biológicos nos quais os fatores de transcrição estão envolvidos:
Regulação transcricional basal
Em eucariotos, uma importante classe de fatores de transcrição chamados fatores de transcrição gerais (GTFs) é necessária para que a transcrição ocorra. Muitos desses GTFs não se ligam realmente ao DNA, mas fazem parte do grande complexo de pré-iniciação da transcrição que interage diretamente com a RNA polimerase. Os GTFs mais comuns são TFIIA, TFIIB, TFIID (ver também proteína de ligação TATA), TFIIE, TFIIF e TFIIH. O complexo de pré-iniciação liga-se a regiões promotoras do DNA a montante do gene que regulam.
Aprimoramento diferencial da transcrição
Outros fatores de transcrição regulam diferencialmente a expressão de vários genes, ligando-se a regiões intensificadoras do DNA adjacentes aos genes regulados. Esses fatores de transcrição são essenciais para garantir que os genes sejam expressos na célula certa, no momento certo e na quantidade certa, dependendo das mudanças nas necessidades do organismo.
Desenvolvimento
Muitos fatores de transcrição em organismos multicelulares estão envolvidos no desenvolvimento. Respondendo a estímulos, estes factores de transcrição ligam/desligam a transcrição dos genes apropriados, o que, por sua vez, permite alterações na morfologia celular ou nas actividades necessárias para a determinação do destino celular e a diferenciação celular. A família do fator de transcrição Hox, por exemplo, é importante para a formação adequada de padrões corporais em organismos tão diversos quanto as moscas da fruta até os humanos. Outro exemplo é o fator de transcrição codificado pelo gene da região Y determinante do sexo (SRY), que desempenha um papel importante na determinação do sexo em humanos.
Did you mean:Response to intracellular signals
As células podem se comunicar entre si liberando moléculas que produzem cascatas de sinalização dentro de outra célula receptora. Se o sinal exigir regulação positiva ou negativa de genes na célula receptora, muitas vezes os fatores de transcrição estarão a jusante na cascata de sinalização. A sinalização de estrogênio é um exemplo de cascata de sinalização bastante curta que envolve o fator de transcrição do receptor de estrogênio: o estrogênio é secretado por tecidos como os ovários e a placenta, atravessa a membrana celular da célula receptora e é ligado ao receptor de estrogênio na célula. #39;s citoplasma. O receptor de estrogênio vai então para o núcleo da célula e se liga aos locais de ligação ao DNA, alterando a regulação transcricional dos genes associados.
Resposta ao meio ambiente
Os fatores de transcrição não apenas atuam a jusante das cascatas de sinalização relacionadas a estímulos biológicos, mas também podem estar a jusante das cascatas de sinalização envolvidas em estímulos ambientais. Os exemplos incluem o fator de choque térmico (HSF), que regula positivamente os genes necessários para a sobrevivência em temperaturas mais altas, o fator induzível por hipóxia (HIF), que regula positivamente os genes necessários para a sobrevivência celular em ambientes com baixo teor de oxigênio, e a proteína de ligação ao elemento regulador de esterol (SREBP), que ajuda a manter os níveis adequados de lipídios na célula.
Controle do ciclo celular
Muitos fatores de transcrição, especialmente alguns que são proto-oncogenes ou supressores de tumor, ajudam a regular o ciclo celular e, como tal, determinam o tamanho que uma célula atingirá e quando poderá se dividir em duas células-filhas. Um exemplo é o oncogene Myc, que desempenha papéis importantes no crescimento celular e na apoptose.
Patogênese
Fatores de transcrição também podem ser usados para alterar a expressão gênica em uma célula hospedeira para promover a patogênese. Um exemplo bem estudado disso são os efetores semelhantes aos ativadores de transcrição (efetores TAL) secretados pela bactéria Xanthomonas. Quando injetadas em plantas, essas proteínas podem entrar no núcleo da célula vegetal, ligar-se a sequências promotoras de plantas e ativar a transcrição de genes vegetais que auxiliam na infecção bacteriana. Os efetores TAL contêm uma região central de repetição na qual existe uma relação simples entre a identidade de dois resíduos críticos em repetições sequenciais e bases sequenciais de DNA no sítio alvo do efetor TAL. Esta propriedade provavelmente facilita a evolução dessas proteínas para competir melhor com os mecanismos de defesa da célula hospedeira.
Regulamento
É comum na biologia que processos importantes tenham múltiplas camadas de regulação e controle. Isto também se aplica aos fatores de transcrição: não apenas os fatores de transcrição controlam as taxas de transcrição para regular as quantidades de produtos gênicos (RNA e proteína) disponíveis para a célula, mas os próprios fatores de transcrição são regulados (muitas vezes por outros fatores de transcrição). Abaixo está uma breve sinopse de algumas das maneiras pelas quais a atividade dos fatores de transcrição pode ser regulada:
Síntese
Fatores de transcrição (como todas as proteínas) são transcritos de um gene em um cromossomo em RNA, e então o RNA é traduzido em proteína. Qualquer uma dessas etapas pode ser regulada para afetar a produção (e, portanto, a atividade) de um fator de transcrição. Uma implicação disso é que os fatores de transcrição podem auto-regular-se. Por exemplo, num ciclo de feedback negativo, o factor de transcrição actua como o seu próprio repressor: se a proteína do factor de transcrição se ligar ao ADN do seu próprio gene, ela regula negativamente a produção de mais de si mesma. Este é um mecanismo para manter níveis baixos de um fator de transcrição em uma célula.
Localização nuclear
Em eucariotos, os fatores de transcrição (como a maioria das proteínas) são transcritos no núcleo, mas depois traduzidos no citoplasma da célula. Muitas proteínas ativas no núcleo contêm sinais de localização nuclear que as direcionam para o núcleo. Mas, para muitos factores de transcrição, este é um ponto chave na sua regulação. Classes importantes de fatores de transcrição, como alguns receptores nucleares, devem primeiro ligar-se a um ligante enquanto estão no citoplasma antes de poderem se deslocar para o núcleo.
Ativação
Os fatores de transcrição podem ser ativados (ou desativados) através de seu domínio de detecção de sinal por vários mecanismos, incluindo:
- ligação ligante – Não só é ligação ligante capaz de influenciar onde um fator de transcrição está localizado dentro de uma célula, mas ligação ligante também pode afetar se o fator de transcrição está em um estado ativo e capaz de ligar DNA ou outros cofactores (veja, por exemplo, receptores nucleares).
- fosforilação – Muitos fatores de transcrição tais como proteínas STAT devem ser fosforilados antes que possam ligar o DNA.
- interação com outros fatores de transcrição (por exemplo., homo- ou hetero-dimerização) ou proteínas coregulatórias
Acessibilidade do local de ligação ao DNA
Nos eucariotos, o DNA é organizado com a ajuda de histonas em partículas compactas chamadas nucleossomos, onde sequências de cerca de 147 pares de bases de DNA dão aproximadamente 1,65 voltas em torno dos octâmeros da proteína histona. O DNA dentro dos nucleossomos é inacessível a muitos fatores de transcrição. Alguns factores de transcrição, os chamados factores pioneiros, ainda são capazes de ligar os seus locais de ligação ao ADN nucleossómico. Para a maioria dos outros fatores de transcrição, o nucleossomo deve ser ativamente desenrolado por motores moleculares, como os remodeladores da cromatina. Alternativamente, o nucleossomo pode ser parcialmente desembrulhado por flutuações térmicas, permitindo acesso temporário ao sítio de ligação do fator de transcrição. Em muitos casos, um fator de transcrição precisa competir pela ligação ao seu sítio de ligação ao DNA com outros fatores de transcrição e histonas ou proteínas da cromatina não-histonas. Pares de fatores de transcrição e outras proteínas podem desempenhar papéis antagônicos (ativador versus repressor) na regulação do mesmo gene.
Disponibilidade de outros cofatores/fatores de transcrição
A maioria dos fatores de transcrição não funciona sozinho. Muitas famílias grandes de TF formam interações homotípicas ou heterotípicas complexas através da dimerização. Para que ocorra a transcrição genética, vários fatores de transcrição devem se ligar às sequências reguladoras do DNA. Esta coleção de fatores de transcrição, por sua vez, recruta proteínas intermediárias, como cofatores, que permitem o recrutamento eficiente do complexo de pré-iniciação e da RNA polimerase. Assim, para que um único factor de transcrição inicie a transcrição, todas estas outras proteínas também devem estar presentes, e o factor de transcrição deve estar num estado em que possa ligar-se a elas, se necessário. Cofatores são proteínas que modulam os efeitos dos fatores de transcrição. Os cofatores são intercambiáveis entre promotores de genes específicos; o complexo proteico que ocupa o DNA promotor e a sequência de aminoácidos do cofator determinam sua conformação espacial. Por exemplo, certos receptores de esteróides podem trocar cofatores com NF-κB, o que é uma troca entre inflamação e diferenciação celular; assim, os esteróides podem afetar a resposta inflamatória e a função de certos tecidos.
Interação com citosina metilada
Fatores de transcrição e citosinas metiladas no DNA têm papéis importantes na regulação da expressão gênica. (A metilação da citosina no DNA ocorre principalmente onde a citosina é seguida pela guanina na sequência 5' a 3' do DNA, um sítio CpG.) A metilação dos sítios CpG em uma região promotora de um gene geralmente reprime a transcrição do gene, enquanto a metilação de CpGs no corpo de um gene aumenta a expressão. As enzimas TET desempenham um papel central na desmetilação de citosinas metiladas. A desmetilação de CpGs num promotor de gene pela actividade da enzima TET aumenta a transcrição do gene.
Os locais de ligação ao DNA de 519 fatores de transcrição foram avaliados. Destes, 169 fatores de transcrição (33%) não tinham dinucleotídeos CpG em seus sítios de ligação, e 33 fatores de transcrição (6%) poderiam se ligar a um motivo contendo CpG, mas não exibiram preferência por um sítio de ligação com um motivo metilado. ou CpG não metilado. Houve 117 fatores de transcrição (23%) que foram inibidos de se ligar à sua sequência de ligação se contivesse um sítio CpG metilado, 175 fatores de transcrição (34%) que tinham ligação aumentada se sua sequência de ligação tivesse um sítio CpG metilado e 25 fatores de transcrição factores (5%) foram inibidos ou tiveram ligação aumentada dependendo de onde na sequência de ligação o CpG metilado estava localizado.
As enzimas TET não se ligam especificamente à metilcitosina, exceto quando recrutadas (ver desmetilação do DNA). Foi demonstrado que múltiplos fatores de transcrição importantes na diferenciação celular e especificação de linhagem, incluindo NANOG, SALL4A, WT1, EBF1, PU.1 e E2A, recrutam enzimas TET para loci genômicos específicos (principalmente intensificadores) para atuar na metilcitosina (mC) e convertê-lo em hidroximetilcitosina hmC (e na maioria dos casos marcá-los para subsequente desmetilação completa em citosina). A conversão de mC em hmC mediada por TET parece interromper a ligação de proteínas de ligação a 5mC, incluindo proteínas MECP2 e MBD (domínio de ligação a metil-CpG), facilitando a remodelação do nucleossomo e a ligação de fatores de transcrição, ativando assim a transcrição desses genes. EGR1 é um importante fator de transcrição na formação da memória. Tem um papel essencial na reprogramação epigenética dos neurônios cerebrais. O fator de transcrição EGR1 recruta a proteína TET1 que inicia uma via de desmetilação do DNA. O EGR1, juntamente com o TET1, é empregado na programação da distribuição de locais de metilação no DNA do cérebro durante o desenvolvimento do cérebro e na aprendizagem (ver Epigenética na aprendizagem e na memória).
Estrutura


Os fatores de transcrição têm estrutura modular e contêm os seguintes domínios:
- Domínio de ligação de ADN (DBD), que se liga a sequências específicas de DNA (enhancer ou promotor. Componente necessário para todos os vetores. Usado para conduzir a transcrição das seqüências do promotor transgene do vetor) adjacente a genes regulados. Seqüências de DNA que ligam fatores de transcrição são muitas vezes referidas como elementos de resposta.
- Domínio de ativação (ANÚNCIO), que contém sítios de ligação para outras proteínas, como os coreguladores de transcrição. Estes sites de ligação são frequentemente referidos como funções de ativação (AFs), Domínio de transactivação (TUDO) ou Domínio de transativação TAD mas não misturar com topologicamente associando o domínio TAD.
- Um opcional domínio de sinalização (SSD)por exemplo., um domínio ligante), que detecta sinais externos e, em resposta, transmite esses sinais para o resto do complexo de transcrição, resultando em up- ou down-regulation da expressão genética. Além disso, os domínios DBD e sinalização podem residir em proteínas separadas que associam dentro do complexo de transcrição para regular a expressão genética.
Domínio de ligação ao DNA
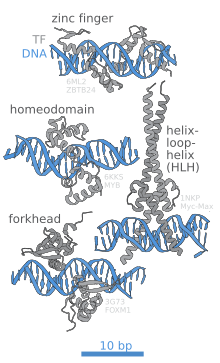
A porção (domínio) do fator de transcrição que se liga ao DNA é chamada de domínio de ligação ao DNA. Abaixo está uma lista parcial de algumas das principais famílias de domínios/fatores de transcrição de ligação ao DNA:
| Família | InterPro | Pfam | SCOP |
|---|---|---|---|
| helix-loop-helix básico | InterPro:IPR001092 | PF00010 | SCOP 47460 |
| zíper básico-leucina (bZIP) | InterPro:IPR004827 | PF00170 | SCOP 57959 |
| Efeito C-terminal ou domínio dos reguladores de resposta bipartida | InterPro:IPR001789 | PF00072 | SCOP 46894 |
| Caixa AP2/ERF/GCC | InterPro:IPR001471 | PF00847 | SCOP 54176 |
| helix-turn-helix | |||
| proteínas homeodomain, que são codificadas por genes homeobox, são fatores de transcrição. As proteínas Homeodomain desempenham papéis críticos na regulação do desenvolvimento. | InterPro:IPR009057 | PF00046 | SCOP 46689 |
| lambda repressor | InterPro:IPR010982 | SCOP 47413 | |
| srf-like (fator de resposta do soro) | InterPro:IPR002100 | PF00319 | SCOP 55455 |
| caixa emparelhada | |||
| hélice alado | InterPro:IPR013196 | PF08279 | SCOP 46785 |
| dedos de zinco | |||
| * multi-domínio Ciclos2O seu2 dedos de zinco | InterPro:IPR007087 | PF00096 | SCOP 57667 |
| Zn2/Cys6 | SCOP 57701 | ||
| Zn2/Cys8 dedo de zinco do receptor nuclear | InterPro:IPR001628 | PF00105 | SCOP 57716 |
Elementos de resposta
A sequência de DNA à qual um fator de transcrição se liga é chamada de sítio de ligação do fator de transcrição ou elemento de resposta.
Os fatores de transcrição interagem com seus locais de ligação usando uma combinação de forças eletrostáticas (das quais as ligações de hidrogênio são um caso especial) e de Van der Waals. Devido à natureza destas interacções químicas, a maioria dos factores de transcrição ligam-se ao ADN de uma forma específica de sequência. No entanto, nem todas as bases no sítio de ligação do fator de transcrição podem realmente interagir com o fator de transcrição. Além disso, algumas destas interações podem ser mais fracas que outras. Assim, os factores de transcrição não se ligam a apenas uma sequência, mas são capazes de se ligar a um subconjunto de sequências estreitamente relacionadas, cada uma com uma força de interacção diferente.
Por exemplo, embora o sítio de ligação de consenso para a proteína de ligação a TATA (TBP) seja TATAAAA, o fator de transcrição TBP também pode ligar sequências semelhantes, como TATATAT ou TATATAA.Como os fatores de transcrição podem se ligar a um conjunto de sequências relacionadas e essas sequências tendem a ser curtas, os potenciais locais de ligação do fator de transcrição podem ocorrer por acaso se a sequência de DNA for longa o suficiente. É improvável, contudo, que um factor de transcrição se ligue a todas as sequências compatíveis no genoma da célula. Outras restrições, como a acessibilidade do DNA na célula ou a disponibilidade de cofatores, também podem ajudar a determinar onde um fator de transcrição realmente se ligará. Assim, dada a sequência do genoma, ainda é difícil prever onde um factor de transcrição se irá realmente ligar numa célula viva.
Especificidade de reconhecimento adicional, entretanto, pode ser obtida através do uso de mais de um domínio de ligação ao DNA (por exemplo, DBDs em tandem no mesmo fator de transcrição ou através da dimerização de dois fatores de transcrição) que se ligam a duas ou mais sequências adjacentes de ADN.
Significado clínico
Os fatores de transcrição têm importância clínica por pelo menos dois motivos: (1) as mutações podem estar associadas a doenças específicas e (2) podem ser alvos de medicamentos.
Transtornos
Devido aos seus importantes papéis no desenvolvimento, na sinalização intercelular e no ciclo celular, algumas doenças humanas têm sido associadas a mutações em fatores de transcrição.
Muitos fatores de transcrição são supressores de tumor ou oncogenes e, portanto, mutações ou regulação aberrante deles estão associadas ao câncer. Sabe-se que três grupos de factores de transcrição são importantes no cancro humano: (1) as famílias NF-kappaB e AP-1, (2) a família STAT e (3) os receptores de esteróides.
Abaixo estão alguns dos exemplos mais bem estudados:
| Condição | Descrição | Locus |
|---|---|---|
| Síndrome de Rett | As mutações no fator de transcrição MECP2 estão associadas à síndrome de Rett, um transtorno neurodesenvolvimento. | Xq28 |
| Diabetes | Uma forma rara de diabetes chamada MODY (diabetes de início de maturidade dos jovens) pode ser causada por mutações em fatores nucleares hepatócitos (HNFs) ou fator promotor de insulina-1 (IPF1/Pdx1). | múltiplo |
| Dispraxia verbal do desenvolvimento | Mutações no fator de transcrição FOXP2 estão associadas à dispraxia verbal do desenvolvimento, uma doença em que os indivíduos são incapazes de produzir os movimentos finamente coordenados necessários para a fala. | 7 |
| Doenças auto-imunes | Mutações no fator de transcrição FOXP3 causam uma forma rara de doença autoimune chamada IPEX. | Xp11.23-q13.3 |
| Síndrome de Li-Fraumeni | Causado por mutações no supressor do tumor p53. | 17p1 |
| Câncer de mama | A família STAT é relevante para câncer de mama. | múltiplo |
| Vários cancros | A família HOX está envolvida em uma variedade de cânceres. | múltiplo |
| Osteoartrite | Mutação ou atividade reduzida de SOX9 |
Potenciais alvos de drogas
Aproximadamente 10% dos medicamentos atualmente prescritos têm como alvo direto a classe de receptores nucleares de fatores de transcrição. Os exemplos incluem tamoxifeno e bicalutamida para o tratamento do câncer de mama e de próstata, respectivamente, e vários tipos de esteróides antiinflamatórios e anabolizantes. Além disso, os fatores de transcrição são frequentemente modulados indiretamente por drogas através de cascatas de sinalização. Pode ser possível atingir diretamente outros fatores de transcrição menos explorados, como o NF-κB, com medicamentos. Acredita-se que os fatores de transcrição fora da família de receptores nucleares sejam mais difíceis de atingir com terapêuticas de moléculas pequenas, uma vez que não está claro se eles são "drogáveis" mas houve progresso no Pax2 e no caminho do entalhe.
Papel na evolução
As duplicações genéticas desempenharam um papel crucial na evolução das espécies. Isto se aplica particularmente aos fatores de transcrição. Uma vez que ocorrem como duplicatas, as mutações acumuladas que codificam para uma cópia podem ocorrer sem afetar negativamente a regulação dos alvos a jusante. No entanto, alterações nas especificidades de ligação ao DNA do fator de transcrição Leafy de cópia única, que ocorre na maioria das plantas terrestres, foram recentemente elucidadas. A esse respeito, um factor de transcrição de cópia única pode sofrer uma mudança de especificidade através de um intermediário promíscuo sem perder função. Mecanismos semelhantes foram propostos no contexto de todas as hipóteses filogenéticas alternativas e no papel dos fatores de transcrição na evolução de todas as espécies.
Papel na atividade de controle biológico
Os fatores de transcrição desempenham um papel na atividade de resistência que é importante para o sucesso da atividade de biocontrole. A resistência ao estresse oxidativo e a detecção de pH alcalino foram contribuídas pelo fator de transcrição Yap1 e Rim101 do Papiliotrema terrestris LS28, pois ferramentas moleculares revelaram uma compreensão dos mecanismos genéticos subjacentes à atividade de biocontrole que apoia programas de gerenciamento de doenças baseados sobre controle biológico e integrado.
Análise
Existem diferentes tecnologias disponíveis para analisar fatores de transcrição. No nível genômico, o sequenciamento de DNA e a pesquisa em bancos de dados são comumente usados. A versão proteica do fator de transcrição é detectável usando anticorpos específicos. A amostra é detectada em um western blot. Ao utilizar o ensaio de mudança de mobilidade eletroforética (EMSA), o perfil de ativação dos fatores de transcrição pode ser detectado. Uma abordagem multiplex para perfil de ativação é um sistema de chip TF onde vários fatores de transcrição diferentes podem ser detectados em paralelo.
O método mais comumente usado para identificar locais de ligação do fator de transcrição é a imunoprecipitação da cromatina (ChIP). Esta técnica baseia-se na fixação química da cromatina com formaldeído, seguida de co-precipitação de DNA e do fator de transcrição de interesse usando um anticorpo que tem como alvo específico essa proteína. As sequências de DNA podem então ser identificadas por microarray ou sequenciamento de alto rendimento (ChIP-seq) para determinar os locais de ligação do fator de transcrição. Se nenhum anticorpo estiver disponível para a proteína de interesse, o DamID pode ser uma alternativa conveniente.
Aulas
Conforme descrito em mais detalhes abaixo, os fatores de transcrição podem ser classificados por seu (1) mecanismo de ação, (2) função regulatória ou (3) homologia de sequência (e, portanto, similaridade estrutural) em seus domínios de ligação ao DNA.
Mecanicista
Existem duas classes mecanísticas de fatores de transcrição:
- Os fatores de transcrição geral estão envolvidos na formação de um complexo de pré-iniciação. Os mais comuns são abreviados como TFIIA, TFIIB, TFIID, TFIIE, TFIIF e TFIIH. Eles são onipresentes e interagem com a região promotora central em torno do site de início de transcrição de todos os genes de classe II.
- Fatores de transcrição a montante são proteínas que se ligam em algum lugar a montante do local de iniciação para estimular ou reprimir a transcrição. Estes são aproximadamente sinônimos fatores de transcrição específicos, porque eles variam consideravelmente dependendo de que sequências de reconhecimento estão presentes na proximidade do gene.
| Exemplos de fatores de transcrição específicos | |||
|---|---|---|---|
| Factor | Tipo estrutural | Sequência de reconhecimento | Ligações como |
| SP1 | Dedo de zinco | 5 'GGGGGG-3' | Monomer |
| AP-1 | Zipper básico | 5'-TGA(G/C)TCA-3 ' | Dimer |
| C/EBP | Zipper básico | 5 'ATTGCGCAAT-3 ' | Dimer |
| Fator de choque térmico | Zipper básico | 5'-XGAAX-3' | Trimer. |
| ATF/CREB | Zipper básico | 5' TGACGTCA-3 ' | Dimer |
| C-Myc | Básico helix-loop-helix | 5'-CACGTG-3' | Dimer |
| O quê? | Helix-turn-helix | 5'-ATGCAAAT-3' | Monomer |
| NF-1 | Romance | 5'-TTGCXXXXXGCCAA-3 ' | Dimer |
| (G/C) = G ou C X = A, T, G ou C | |||
Funcional
Os fatores de transcrição foram classificados de acordo com sua função reguladora:
- Eu. constitutiva activa – presente em todas as células em todos os momentos – fatores de transcrição geral, Sp1, NF1, CCAAT
- II. condicionalmente ativo – requer ativação
- II.A desenvolvimento (especificidade da célula) – a expressão é controlada firmemente, mas, uma vez expressa, não requer ativação adicional – GATA, HNF, PIT-1, MyoD, Myf5, Hox, Winged Helix
- II.B dependente do sinal – requer sinal externo para ativação
- II.B.1 ligante extracelular (endocrino ou paracrino)-dependente – receptores nucleares
- II.B.2 ligante intracelular (autocrino)-dependente – ativado por pequenas moléculas intracelulares – SREBP, p53, receptores nucleares órfãos
- II.B.3 receptor de membrana celular dependente – segundo mensageiro sinalização cascatas resultando na fosforilação do fator de transcrição
- II.B.3.a fatores nucleares residentes – residir no núcleo, independentemente do estado de ativação – CREB, AP-1, Mef2
- II.B.3.b fatores citoplasmáticos latentes – forma inativa reside no citoplasma, mas, quando ativado, são translocados no núcleo – STAT, R-SMAD, NF-κB, Notch, TUBBY, NFAT
Estrutural
Os fatores de transcrição são frequentemente classificados com base na similaridade de sequência e, portanto, na estrutura terciária de seus domínios de ligação ao DNA:
- 1 Superclasse: Domínios Básicos
- 1.1.1. Classe: Fatores de zíper leucina (bZIP)
- 1.1.1 Família: componentes AP-1(-como); inclui (c-Fos/c-Jun)
- 1.1.2 Família: CREB
- 1.1.3 Família: C/EBP-como fatores
- 1.1.4 Família: bZIP / PAR
- 1.1.5 Família: Planta G-box fatores de ligação
- 1.1.6 Família: ZIP somente
- 1.2. Classe: Fatores Helix-loop-helix (bHLH)
- 1.2.1 Família: Fatores Ubiquitous (classe A)
- 1.2.2 Família: Fatores de transcrição miogênica (MyoD)
- 1.2.3 Família: Achaete-Scute
- 1.2.4 Família: Tal/Twist/Atonal/Hen
- 1.3. Classe: Helix-loop-helix / fatores de zíper leucina (bHLH-ZIP)
- 1.3.1 Família: Ubiquitous bHLH-ZIP fatores; inclui USF (USF1, USF2); SREBP (SREBP)
- 1.3.2 Família: Fatores de controle de ciclo celular; inclui c-Myc
- UNIÃO EUROPEIA Classe: NF-1
- 1.4.1 Família: NF-1 (A, B, C, X)
- 1.5. Classe: RF-X
- 1.5.1 Família: RF-X (1, 2, 3, 4, 5, ANK)
- 1.6 Classe: BHSH
- 1.1.1. Classe: Fatores de zíper leucina (bZIP)
- 2 Superclasse: Zinco-coordenando domínios de ligação de DNA
- 2. Classe: Dedo de zinco Cys4 do tipo receptor nuclear
- 2.1.1 Família: Receptores hormonais esteroides
- 2.1.2 Família: Fatores semelhantes ao receptor do hormônio tireóide
- 2.2 Classe: diversos dedos de zinco Cys4
- 2.2.1 Família: GATA-Factores
- 2.3 Classe: Cys2 Domínio de dedos de zinco His2
- 2.3.1 Família: Fatores ubíquos, inclui TFIIIA, Sp1
- 2.3.2 Família: Reguladores do ciclo de desenvolvimento/célula; inclui Krüppel
- 2.3.4 Família: Grandes fatores com propriedades de ligação tipo NF-6B
- 2. Classe: Cisteína-zinc cluster
- 2.5. Classe: Dedos de zinco de composição alternada
- 2. Classe: Dedo de zinco Cys4 do tipo receptor nuclear
- 3 Superclasse: Helix-turn-helix
- 3.1 Classe: Domínio de Homeo
- 3.1.1 Família: domínio homeo somente; inclui Ubx
- 3.1.2 Família: fatores de domínio POU; inclui Oct
- 3.1.3 Família: Domínio homeo com região LIM
- 3.1.4 Família: domínio homeo mais motivos do dedo de zinco
- 3.2. Classe: Caixa emparelhada
- 3.2.1 Família: Emparelhado mais domínio homeo
- 3.2.2 Família: Apenas domínio pareado
- 3.3 Classe: Cabeça de garfo / hélice asa
- 3.3.1 Família: Reguladores do desenvolvimento; inclui cabeça de garfo
- 3.3.2 Família: Reguladores específicos do tecido
- 3.3.3 Família: Fatores de controle do ciclo celular
- 3.3.0 Família: Outros reguladores
- 3.4 Classe: Fatores de choque térmico
- 3.4.1 Família: HSF
- 3.5 Classe: Aglomerados triptofanos
- Família: Myb
- Família 3.5.2: Tipo de Ets
- 3.5.3 Família: Fatores regulatórios interferon
- 3.6 Classe: domínio TEA (factor de potenciador de assinatura)
- 3.6.1 Família: TEA (TEAD1, TEAD2, TEAD3, TEAD4)
- 3.1 Classe: Domínio de Homeo
- 4 Superclasse: beta-Scaffold Fatores com contatos menores do sulco
- 4.1 Classe: RH (Rel homology region)
- 4.1.1 Família: Rel/ankyrin; NF-kappaB
- 4.1.2 Família: apenas ankyrin
- 4.1.3 Família: NFAT (N- Claro. Fator de Activado T-células) (NFATC1, NFATC2, NFATC3)
- - Sim. Classe: ESTATA
- 4.2.1 Família: STAT
- 4.3 Classe: p53
- 4.3.1 Família: p53
- 4.4 Classe: caixa MADS
- 4.4.1 Família: Reguladores de diferenciação; inclui (Mef2)
- 4.4.2 Família: Respondentes a sinais externos, SRF (fator de resposta do soro) (SRF)
- 4.4.3 Família: Reguladores Metabólicos (ARG80)
- 4.5 Classe: fatores de transcrição alfa-helix beta-Barrel
- 4.6 Classe: Proteínas de ligação TATA
- 4.6.1 Família: TBP
- 4.7. Classe: HMG-box
- 4.7.1 Família: genes SOX, SRY
- 4.7.2 Família: TCF-1 (TCF1)
- 4.7.3 Família: HMG2 relacionada, SSRP1
- 4.7.4 Família: UBF
- 4.7.5 Família: MATA
- 4.8 Classe: Fatores CCAAT heteroméricos
- 4.8.1 Família: Heteromeric CCAAT fatores
- 4.9 Classe: Cabeça de grão
- 4.9.1 Família: Grainyhead
- 4.10 Classe: Fatores de domínio Cold-shock
- 4.10.1 Família: csd
- 4.11. Classe: Runt
- 4.11.1 Família: Runt
- 4.1 Classe: RH (Rel homology region)
- 0 Superclasse: Outros Fatores de Transcrição
- 0.1 Classe: Proteínas de punho de cobre
- 0.2 classe: HMGI(Y) (HMGA1)
- 0.2.1 Família: HMGI(Y)
- 0 Classe: Domínio de bolso
- 0 Classe: E1A-como fatores
- 0,5 Classe: Fatores relacionados ao AP2/EREBP
- Família 0.5.1: AP2
- Família: EREBP
- 0,5.3 Superfamília: AP2/B3
- Família: ARF
- 0,5.3.2 Família: ABI
- 0,5.3.3 Família: RAV
Bancos de dados de fatores de transcrição
Existem vários bancos de dados que catalogam informações sobre fatores de transcrição, mas seu escopo e utilidade variam dramaticamente. Alguns podem conter apenas informações sobre as proteínas reais, alguns sobre os seus locais de ligação ou sobre os seus genes alvo. Os exemplos incluem o seguinte:
- pegada DB-- uma metadadosbase de múltiplas bases de dados, incluindo JASPAR e outros
- JASPAR: banco de dados de sites de ligação de fator de transcrição para eukaryotes
- PlantTFD: Base de dados de fator de transcrição vegetal
- TcoF-DB: Banco de dados de co-factores de transcrição e interações do fator de transcrição
- TFcheckpoint: banco de dados de candidatos humanos, ratos e ratos TF
- transcriptionfactor.org (agora comercial, vendendo reagentes)
Contenido relacionado
Alexandre Fleming
Emil Theodor Kocher
Kary Mullis
Lactase
Konrad Lorenz