Estrutura terciária da proteína

Estrutura terciária da proteína é a forma tridimensional de uma proteína. A estrutura terciária terá uma única cadeia polipeptídica "espinha dorsal" com uma ou mais estruturas secundárias de proteínas, os domínios de proteínas. As cadeias laterais de aminoácidos podem interagir e se ligar de várias maneiras. As interações e ligações das cadeias laterais dentro de uma determinada proteína determinam sua estrutura terciária. A estrutura terciária da proteína é definida por suas coordenadas atômicas. Essas coordenadas podem se referir a um domínio de proteína ou a toda a estrutura terciária. Várias estruturas terciárias podem se dobrar em uma estrutura quaternária.
História
A ciência da estrutura terciária das proteínas progrediu de uma hipótese para uma definição detalhada. Embora Emil Fischer tivesse sugerido que as proteínas eram feitas de cadeias polipeptídicas e cadeias laterais de aminoácidos, foi Dorothy Maud Wrinch quem incorporou a geometria à previsão das estruturas proteicas. Wrinch demonstrou isso com o modelo Cyclol, a primeira previsão da estrutura de uma proteína globular. Os métodos contemporâneos são capazes de determinar, sem previsão, estruturas terciárias dentro de 5 Å (0,5 nm) para proteínas pequenas (<120 resíduos) e, sob condições favoráveis, previsões confiáveis de estruturas secundárias.
Determinantes
Estabilidade dos estados nativos
Termoestabilidade
Uma proteína dobrada em seu estado nativo ou conformação nativa normalmente tem uma energia livre de Gibbs mais baixa (uma combinação de entalpia e entropia) do que a conformação desdobrada. Uma proteína tenderá a conformações de baixa energia, o que determinará o enovelamento da proteína no meio celular. Como muitas conformações semelhantes terão energias semelhantes, as estruturas das proteínas são dinâmicas, flutuando entre essas estruturas semelhantes.
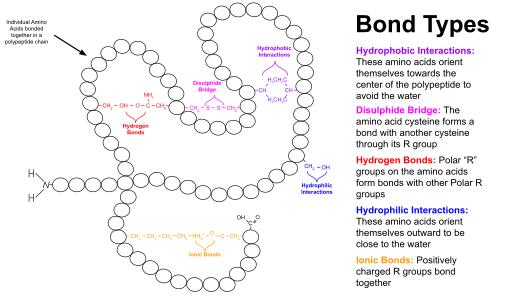
As proteínas globulares têm um núcleo de resíduos de aminoácidos hidrofóbicos e uma região de superfície de resíduos hidrofílicos, carregados e expostos à água. Este arranjo pode estabilizar as interações dentro da estrutura terciária. Por exemplo, nas proteínas secretadas, que não são banhadas pelo citoplasma, as pontes dissulfeto entre os resíduos de cisteína ajudam a manter a estrutura terciária. Existe uma semelhança de estruturas terciárias estáveis observadas em proteínas de diversas funções e diversas evoluções. Por exemplo, o barril TIM, nomeado para a enzima triosefosfatoisomerase, é uma estrutura terciária comum, assim como a estrutura espiral dimérica altamente estável. Assim, as proteínas podem ser classificadas pelas estruturas que possuem. Bancos de dados de proteínas que usam essa classificação incluem SCOP e CATH.
Armadilhas cinéticas
A cinética de dobramento pode prender uma proteína em uma conformação de alta energia, ou seja, uma conformação intermediária de alta energia bloqueia o acesso à conformação de energia mais baixa. A conformação de alta energia pode contribuir para a função da proteína. Por exemplo, a proteína hemaglutinina da gripe é uma única cadeia polipeptídica que, quando ativada, é clivada proteoliticamente para formar duas cadeias polipeptídicas. As duas cadeias são mantidas em uma conformação de alta energia. Quando o pH local cai, a proteína sofre um rearranjo conformacional energeticamente favorável que permite que ela penetre na membrana da célula hospedeira.
Metabilidade
Algumas estruturas proteicas terciárias podem existir em estados de vida longa que não são o estado mais estável esperado. Por exemplo, muitas serpinas (inibidores de serina protease) apresentam essa metaestabilidade. Eles sofrem uma mudança conformacional quando um loop da proteína é cortado por uma protease.
Proteínas acompanhantes
É comumente assumido que o estado nativo de uma proteína também é o mais termodinamicamente estável e que uma proteína atingirá seu estado nativo, dada sua cinética química, antes de ser traduzida. As chaperonas proteicas no citoplasma de uma célula auxiliam um polipeptídeo recém-sintetizado a atingir seu estado nativo. Algumas proteínas acompanhantes são altamente específicas em sua função, por exemplo, proteína dissulfeto isomerase; outras são gerais em sua função e podem auxiliar a maioria das proteínas globulares, por exemplo, o sistema de proteínas procarióticas GroEL/GroES e as proteínas de choque térmico eucarióticas homólogas (o sistema Hsp60/Hsp10).
Ambiente citoplasmático
A previsão da estrutura terciária da proteína depende do conhecimento da estrutura primária da proteína e da comparação da possível estrutura terciária prevista com estruturas terciárias conhecidas em bancos de dados de proteínas. Isso leva em consideração apenas o ambiente citoplasmático presente no momento da síntese de proteínas, na medida em que um ambiente citoplasmático semelhante também pode ter influenciado a estrutura das proteínas registradas no banco de dados de proteínas.
Ligação do ligante
A estrutura de uma proteína, por exemplo, uma enzima, pode mudar após a ligação de seus ligantes naturais, por exemplo, um cofator. Neste caso, a estrutura da proteína ligada ao ligante é conhecida como estrutura holo, da proteína não ligada como estrutura apo.
Estrutura estabilizada pela formação de ligações fracas entre cadeias laterais de aminoácidos - Determinado pelo dobramento da cadeia polipeptídica sobre si mesma (resíduos apolares estão localizados dentro da proteína, enquanto os resíduos polares estão localizados principalmente fora) - Envelopamento da proteína aproxima a proteína e relaciona a-a localizados em regiões distantes da sequência - A aquisição da estrutura terciária leva à formação de bolsões e locais adequados para o reconhecimento e a ligação de moléculas específicas (bioespecificidade).
Determinação
O conhecimento da estrutura terciária das proteínas globulares solúveis é mais avançado do que o das proteínas de membrana porque as primeiras são mais fáceis de estudar com a tecnologia disponível.
Cristalografia de raios-X
A cristalografia de raios-X é a ferramenta mais comum usada para determinar a estrutura da proteína. Ele fornece alta resolução da estrutura, mas não fornece informações sobre a flexibilidade conformacional da proteína.
RMN
Proteína NMR dá resolução comparativamente menor da estrutura da proteína. É limitado a proteínas menores. No entanto, pode fornecer informações sobre alterações conformacionais de uma proteína em solução.
Microscopia eletrônica criogênica
A microscopia eletrônica criogênica (cryo-EM) pode fornecer informações sobre a estrutura terciária e quaternária de uma proteína. É particularmente adequado para grandes proteínas e complexos simétricos de subunidades de proteínas.
Interferometria de dupla polarização
A interferometria de dupla polarização fornece informações complementares sobre as proteínas capturadas na superfície. Auxilia na determinação de mudanças de estrutura e conformação ao longo do tempo.
Projetos
Algoritmo de previsão
O projeto Folding@home da Universidade de Stanford é um esforço de pesquisa de computação distribuída que usa aproximadamente 5 petaFLOPS (≈10 x86 petaFLOPS) de computação disponível. O objetivo é encontrar um algoritmo que preveja consistentemente as estruturas terciárias e quaternárias das proteínas, dada a sequência de aminoácidos da proteína e suas condições celulares.
Uma lista de software para previsão da estrutura terciária de proteínas pode ser encontrada em Lista de software de previsão de estrutura de proteínas.
Doenças de agregação de proteínas
Doenças de agregação de proteínas, como a doença de Alzheimer e a doença de Huntington, e doenças causadas por príons, como a encefalopatia espongiforme bovina, podem ser melhor compreendidas construindo (e reconstruindo) modelos de doenças. Isso é feito causando a doença em animais de laboratório, por exemplo, pela administração de uma toxina, como o MPTP para causar a doença de Parkinson, ou por meio de manipulação genética. A previsão da estrutura de proteínas é uma nova forma de criar modelos de doenças, o que pode evitar o uso de animais.
Projeto de Recuperação de Estrutura Terciária de Proteína (CoMOGrad)
Combinar padrões na estrutura terciária de uma determinada proteína com um grande número de estruturas terciárias de proteínas conhecidas e recuperar as mais semelhantes em ordem de classificação está no centro de muitas áreas de pesquisa, como previsão de função de novas proteínas, estudo da evolução, diagnóstico de doenças, descoberta de drogas, design de anticorpos, etc. O projeto CoMOGrad na BUET é um esforço de pesquisa para criar um método extremamente rápido e muito preciso para a recuperação da estrutura terciária de proteínas e desenvolver uma ferramenta on-line com base nos resultados da pesquisa.