Biossíntese de proteínas
Biossíntese de proteínas (ou síntese de proteínas) é um processo biológico central, que ocorre dentro das células, equilibrando a perda de proteínas celulares (via degradação ou exportação) por meio da produção de novas proteínas. As proteínas desempenham várias funções críticas como enzimas, proteínas estruturais ou hormônios. A síntese de proteínas é um processo muito semelhante para procariontes e eucariotos, mas existem algumas diferenças distintas.
A síntese de proteínas pode ser dividida amplamente em duas fases - transcrição e tradução. Durante a transcrição, uma seção do DNA que codifica uma proteína, conhecida como gene, é convertida em uma molécula modelo chamada RNA mensageiro (mRNA). Essa conversão é realizada por enzimas, conhecidas como RNA polimerases, no núcleo da célula. Em eucariotos, este mRNA é inicialmente produzido de forma prematura (pré-mRNA) que sofre modificações pós-transcricionais para produzir mRNA maduro. O mRNA maduro é exportado do núcleo da célula através dos poros nucleares para o citoplasma da célula para que ocorra a tradução. Durante a tradução, o mRNA é lido por ribossomos que usam a sequência de nucleotídeos do mRNA para determinar a sequência de aminoácidos. Os ribossomos catalisam a formação de ligações peptídicas covalentes entre os aminoácidos codificados para formar uma cadeia polipeptídica.
Após a tradução, a cadeia polipeptídica deve se dobrar para formar uma proteína funcional; por exemplo, para funcionar como uma enzima, a cadeia polipeptídica deve se dobrar corretamente para produzir um sítio ativo funcional. Para adotar uma forma tridimensional (3D) funcional, a cadeia polipeptídica deve primeiro formar uma série de estruturas subjacentes menores chamadas estruturas secundárias. A cadeia polipeptídica nessas estruturas secundárias então se dobra para produzir a estrutura terciária 3D geral. Uma vez dobrada corretamente, a proteína pode sofrer mais maturação por meio de diferentes modificações pós-traducionais. Modificações pós-traducionais podem alterar a capacidade da proteína de funcionar, onde ela está localizada dentro da célula (por exemplo, citoplasma ou núcleo) e a capacidade da proteína de interagir com outras proteínas.
A biossíntese de proteínas tem um papel fundamental na doença, pois alterações e erros nesse processo, por meio de mutações subjacentes no DNA ou dobramento incorreto de proteínas, geralmente são as causas subjacentes de uma doença. As mutações do DNA alteram a sequência de mRNA subsequente, que então altera a sequência de aminoácidos codificada pelo mRNA. As mutações podem fazer com que a cadeia polipeptídica seja mais curta, gerando uma sequência de parada que causa o término precoce da tradução. Alternativamente, uma mutação na sequência do mRNA altera o aminoácido específico codificado naquela posição na cadeia polipeptídica. Essa alteração de aminoácidos pode afetar a capacidade da proteína de funcionar ou se dobrar corretamente. Proteínas mal dobradas são frequentemente implicadas em doenças, pois as proteínas mal dobradas têm a tendência de se unir para formar aglomerados densos de proteínas. Esses aglomerados estão ligados a uma série de doenças, muitas vezes neurológicas, incluindo a doença de Alzheimer e a doença de Parkinson.
Transcrição
A transcrição ocorre no núcleo usando o DNA como modelo para produzir o mRNA. Em eucariotos, essa molécula de mRNA é conhecida como pré-mRNA, pois sofre modificações pós-transcricionais no núcleo para produzir uma molécula de mRNA madura. No entanto, em procariotos, modificações pós-transcricionais não são necessárias, de modo que a molécula de mRNA madura é imediatamente produzida por transcrição.
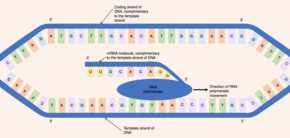
Inicialmente, uma enzima conhecida como helicase atua na molécula do DNA. O DNA possui uma estrutura antiparalela e dupla hélice composta por dois fios de polinucleotídeos complementares, mantidos juntos por ligações de hidrogênio entre os pares de bases. A helicase interrompe as ligações de hidrogênio que causam uma região de DNA - correspondendo a um gene - para relaxar, separando os dois fios de DNA e expondo uma série de bases. Apesar de o DNA ser uma molécula de fita dupla, apenas um dos fios atua como um modelo para a síntese pré-mRNA-essa fita é conhecida como fita de modelo. A outra fita de DNA (que é complementar à fita de modelo) é conhecida como fita de codificação.
Tanto o DNA quanto o RNA têm direcionalidade intrínseca, o que significa que existem duas extremidades distintas da molécula. Essa propriedade da direcionalidade se deve às subunidades de nucleotídeos subjacentes assimétricas, com um grupo fosfato em um lado do açúcar pentose e uma base na outra. Os cinco carbonos no açúcar pentose são numerados de 1 ' (onde ' significa primo) a 5 '. Portanto, as ligações fosfodiestres que conectam os nucleotídeos são formadas juntando -se ao grupo hidroxila no 3 ' carbono de um nucleotídeo para o grupo fosfato nos 5 ' Carbono de outro nucleotídeo. Portanto, a cadeia de codificação do DNA é executada em um 5 ' a 3 ' A direção e a fita de DNA complementares e complementares corre na direção oposta de 3 ' a 5 '.
A enzima RNA polimerase se liga à fita de modelo exposta e lê do gene no 3 ' a 5 ' direção. Simultaneamente, a RNA polimerase sintetiza uma única fita de pré-mRNA no 5 '-39; Direção catalisando a formação de ligações de fosfodiester entre nucleotídeos ativados (livre no núcleo) que são capazes de emparelhamento de bases complementares com a fita de modelo. Por trás da RNA em movimento, os dois fios de DNA se juntam, portanto, apenas 12 pares de Base de DNA são expostos ao mesmo tempo. A RNA polimerase constrói a molécula pré-mRNA a uma taxa de 20 nucleotídeos por segundo, permitindo a produção de milhares de moléculas pré-mRNA do mesmo gene em uma hora. Apesar da rápida taxa de síntese, a enzima RNA polimerase contém seu próprio mecanismo de revisão. Os mecanismos de revisão permitem que a RNA polimerase remova os nucleotídeos incorretos (que não são complementares à fita de DNA do modelo) da crescente molécula pré-mRNA através de uma reação de excisão. Quando as polimerases de RNA atingem uma sequência de DNA específica que termina a transcrição, a RNA polimerase se destaca e a síntese de pré-mRNA está completa.
A molécula pré-mRNA sintetizada é complementar à fita de DNA do modelo e compartilha a mesma sequência de nucleotídeos que a cadeia de DNA de codificação. No entanto, há uma diferença crucial na composição nucleotídica das moléculas de DNA e mRNA. O DNA é composto pelas bases - guanina, citosina, adenina e timina (G, C, A e T) - RNA também é composto por quatro bases - guanina, citosina, adenina e uracil. Nas moléculas de RNA, a timina da base de DNA é substituída pelo uracil, capaz de basear o par com a adenina. Portanto, na molécula pré-mRNA, todas as bases complementares que seriam timina na fita de DNA de codificação são substituídas por uracil.
Modificações pós-transcricionais
Depois que a transcrição estiver concluída, a molécula pré-mRNA passa por modificações pós-transcricionais para produzir uma molécula de mRNA madura.
Existem 3 etapas principais nas modificações pós-transcricionais:
- Adição de uma tampa de 5 ' para a extremidade 5 ' da molécula pré-mRNA
- A adição de uma cauda poli(A) de 3 ' é adicionada à molécula pré-mRNA final de 3 '
- Remoção de introns via splicing RNA
Os 5' boné é adicionado ao 5' final da molécula de pré-mRNA e é composto por um nucleotídeo guanina modificado por metilação. O objetivo do 5' cap serve para evitar a quebra de moléculas de mRNA maduras antes da tradução, o cap também auxilia na ligação do ribossomo ao mRNA para iniciar a tradução e permite que o mRNA seja diferenciado de outros RNAs na célula. Em contraste, o 3' A cauda Poly(A) é adicionada ao 3' final da molécula de mRNA e é composto por 100-200 bases de adenina. Essas modificações distintas de mRNA permitem que a célula detecte que a mensagem completa de mRNA está intacta se ambas as células 5' boné e 3' cauda estão presentes.
Essa molécula de pré-mRNA modificada passa então pelo processo de splicing de RNA. Os genes são compostos de uma série de íntrons e éxons, os íntrons são sequências de nucleotídeos que não codificam uma proteína, enquanto os éxons são sequências de nucleotídeos que codificam diretamente uma proteína. Íntrons e éxons estão presentes tanto na sequência de DNA subjacente quanto na molécula de pré-mRNA, portanto, para produzir uma molécula de mRNA madura que codifica uma proteína, o splicing deve ocorrer. Durante o splicing, os íntrons intervenientes são removidos da molécula de pré-mRNA por um complexo multiproteico conhecido como spliceossoma (composto por mais de 150 proteínas e RNA). Esta molécula de mRNA madura é então exportada para o citoplasma através de poros nucleares no envelope do núcleo.
Tradução

Durante a tradução, os ribossomos sintetizam cadeias polipeptídicas a partir de moléculas modelo de mRNA. Nos eucariotos, a tradução ocorre no citoplasma da célula, onde os ribossomos estão localizados flutuando livremente ou ligados ao retículo endoplasmático. Nos procariotos, que não possuem núcleo, os processos de transcrição e tradução ocorrem no citoplasma.
Os ribossomos são máquinas moleculares complexas, formadas por uma mistura de proteínas e RNA ribossômico, arranjadas em duas subunidades (uma grande e outra pequena), que envolvem a molécula de mRNA. O ribossomo lê a molécula de mRNA em um 5'-3' direção e a utiliza como modelo para determinar a ordem dos aminoácidos na cadeia polipeptídica. Para traduzir a molécula de mRNA, o ribossomo usa pequenas moléculas, conhecidas como RNAs de transferência (tRNA), para entregar os aminoácidos corretos ao ribossomo. Cada tRNA é composto de 70-80 nucleotídeos e adota uma estrutura característica de trevo devido à formação de pontes de hidrogênio entre os nucleotídeos dentro da molécula. Existem cerca de 60 tipos diferentes de tRNAs, cada tRNA se liga a uma sequência específica de três nucleotídeos (conhecida como códon) dentro da molécula de mRNA e entrega um aminoácido específico.
O ribossomo inicialmente se liga ao mRNA no códon de início (AUG) e começa a traduzir a molécula. A sequência de nucleotídeos do mRNA é lida em tripletos - três nucleotídeos adjacentes na molécula de mRNA correspondem a um único códon. Cada tRNA possui uma sequência exposta de três nucleotídeos, conhecida como anticódon, que são complementares em sequência a um códon específico que pode estar presente no mRNA. Por exemplo, o primeiro códon encontrado é o códon de iniciação composto pelos nucleotídeos AUG. O tRNA correto com o anticódon (sequência complementar de 3 nucleotídeos UAC) se liga ao mRNA usando o ribossomo. Este tRNA entrega o aminoácido correto correspondente ao códon do mRNA, no caso do códon de início, este é o aminoácido metionina. O próximo códon (adjacente ao códon de iniciação) é então ligado pelo tRNA correto com anticódon complementar, entregando o próximo aminoácido ao ribossomo. O ribossomo então usa sua atividade enzimática de peptidil transferase para catalisar a formação da ligação peptídica covalente entre os dois aminoácidos adjacentes.
O ribossomo então se move ao longo da molécula de mRNA para o terceiro códon. O ribossomo então libera a primeira molécula de tRNA, pois apenas duas moléculas de tRNA podem ser reunidas por um único ribossomo de cada vez. O próximo tRNA complementar com o anticódon correto complementar ao terceiro códon é selecionado, entregando o próximo aminoácido ao ribossomo que é ligado covalentemente à cadeia polipeptídica em crescimento. Esse processo continua com o ribossomo se movendo ao longo da molécula de mRNA adicionando até 15 aminoácidos por segundo à cadeia polipeptídica. Atrás do primeiro ribossomo, até 50 ribossomos adicionais podem se ligar à molécula de mRNA formando um polissomo, o que permite a síntese simultânea de múltiplas cadeias polipeptídicas idênticas. A terminação da cadeia polipeptídica crescente ocorre quando o ribossomo encontra um códon de parada (UAA, UAG ou UGA) na molécula de mRNA. Quando isso ocorre, nenhum tRNA pode reconhecê-lo e um fator de liberação induz a liberação da cadeia polipeptídica completa do ribossomo. Dr. Har Gobind Khorana, um cientista originário da Índia, decodificou as seqüências de RNA para cerca de 20 aminoácidos. Ele recebeu o prêmio Nobel em 1968, junto com outros dois cientistas, por seu trabalho.
Dobramento de proteínas
Uma vez que a síntese da cadeia polipeptídica é concluída, a cadeia polipeptídica se dobra para adotar uma estrutura específica que permite que a proteína desempenhe suas funções. A forma básica da estrutura da proteína é conhecida como estrutura primária, que é simplesmente a cadeia polipeptídica, ou seja, uma sequência de aminoácidos ligados covalentemente. A estrutura primária de uma proteína é codificada por um gene. Portanto, qualquer alteração na sequência do gene pode alterar a estrutura primária da proteína e todos os níveis subsequentes da estrutura da proteína, alterando, em última análise, a estrutura e a função geral.
A estrutura primária de uma proteína (a cadeia polipeptídica) pode então se dobrar ou enrolar para formar a estrutura secundária da proteína. Os tipos mais comuns de estrutura secundária são conhecidos como hélice alfa ou folha beta, são pequenas estruturas produzidas por pontes de hidrogênio que se formam dentro da cadeia polipeptídica. Essa estrutura secundária então se dobra para produzir a estrutura terciária da proteína. A estrutura terciária é a estrutura 3D geral das proteínas, que é feita de diferentes estruturas secundárias que se dobram juntas. Na estrutura terciária, as principais características da proteína, e. o sítio ativo, são dobrados e formados permitindo que a proteína funcione. Finalmente, algumas proteínas podem adotar uma estrutura quaternária complexa. A maioria das proteínas é composta por uma única cadeia polipeptídica, no entanto, algumas proteínas são compostas por múltiplas cadeias polipeptídicas (conhecidas como subunidades) que se dobram e interagem para formar a estrutura quaternária. Assim, a proteína global é um complexo de subunidades múltiplas composto por subunidades de cadeia polipeptídica dobradas múltiplas, e. hemoglobina.
Eventos pós-tradução
Existem eventos que seguem a biossíntese de proteínas, como proteólise e dobramento de proteínas. A proteólise refere-se à clivagem de proteínas por proteases e à quebra de proteínas em aminoácidos pela ação de enzimas.
Modificações pós-tradução
Quando a dobragem da proteína no estado 3D funcional e maduro é concluída, não é necessariamente o fim da via de maturação da proteína. Uma proteína dobrada ainda pode sofrer processamento adicional por meio de modificações pós-traducionais. Existem mais de 200 tipos conhecidos de modificação pós-traducional, essas modificações podem alterar a atividade da proteína, a capacidade da proteína de interagir com outras proteínas e onde a proteína é encontrada dentro da célula, por exemplo. no núcleo ou no citoplasma da célula. Por meio de modificações pós-traducionais, a diversidade de proteínas codificadas pelo genoma é expandida em 2 a 3 ordens de grandeza.
Existem quatro classes principais de modificação pós-traducional:
- Escavação
- Adição de grupos químicos
- Adição de moléculas complexas
- Formação de obrigações intramoleculares
Decote
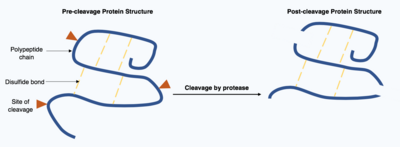
A clivagem de proteínas é uma modificação pós-traducional irreversível realizada por enzimas conhecidas como proteases. Essas proteases geralmente são altamente específicas e causam a hidrólise de um número limitado de ligações peptídicas na proteína-alvo. A proteína encurtada resultante tem uma cadeia polipeptídica alterada com diferentes aminoácidos no início e no final da cadeia. Essa modificação pós-traducional frequentemente altera a função das proteínas, a proteína pode ser inativada ou ativada pela clivagem e pode exibir novas atividades biológicas.
Adição de grupos químicos
Após a tradução, pequenos grupos químicos podem ser adicionados aos aminoácidos dentro da estrutura da proteína madura. Exemplos de processos que adicionam grupos químicos à proteína alvo incluem metilação, acetilação e fosforilação.
Metilação é a adição reversível de um grupo metil em um aminoácido catalisada por enzimas metiltransferase. A metilação ocorre em pelo menos 9 dos 20 aminoácidos comuns, no entanto, ocorre principalmente nos aminoácidos lisina e arginina. Um exemplo de uma proteína que é comumente metilada é uma histona. As histonas são proteínas encontradas no núcleo da célula. O DNA é firmemente enrolado em torno das histonas e mantido no lugar por outras proteínas e interações entre cargas negativas no DNA e cargas positivas na histona. Um padrão altamente específico de metilação de aminoácidos nas proteínas histonas é usado para determinar quais regiões do DNA são fortemente enroladas e incapazes de serem transcritas e quais regiões são frouxamente enroladas e capazes de serem transcritas.
A regulação baseada em histonas da transcrição do DNA também é modificada pela acetilação. A acetilação é a adição covalente reversível de um grupo acetil a um aminoácido lisina pela enzima acetiltransferase. O grupo acetil é removido de uma molécula doadora conhecida como acetil coenzima A e transferido para a proteína alvo. As histonas sofrem acetilação em seus resíduos de lisina por enzimas conhecidas como histona acetiltransferase. O efeito da acetilação é enfraquecer as interações de carga entre a histona e o DNA, tornando assim mais genes no DNA acessíveis para transcrição.
A modificação final e prevalente do grupo químico pós-traducional é a fosforilação. A fosforilação é a adição covalente e reversível de um grupo fosfato a aminoácidos específicos (serina, treonina e tirosina) dentro da proteína. O grupo fosfato é removido da molécula doadora de ATP por uma proteína quinase e transferido para o grupo hidroxila do aminoácido alvo, produzindo difosfato de adenosina como um subproduto. Esse processo pode ser revertido e o grupo fosfato removido pela enzima proteína fosfatase. A fosforilação pode criar um sítio de ligação na proteína fosforilada que permite que ela interaja com outras proteínas e gere grandes complexos multiproteicos. Alternativamente, a fosforilação pode alterar o nível de atividade da proteína, alterando a capacidade da proteína de se ligar ao seu substrato.
Adição de moléculas complexas
As modificações pós-traducionais podem incorporar moléculas grandes e mais complexas na estrutura da proteína dobrada. Um exemplo comum disso é a glicosilação, a adição de uma molécula de polissacarídeo, que é amplamente considerada a modificação pós-traducional mais comum.
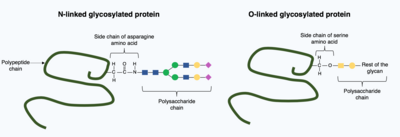
Na glicosilação, uma molécula de polissacarídeo (conhecida como glicano) é adicionada covalentemente à proteína alvo por enzimas glicosiltransferases e modificada por glicosidases no retículo endoplasmático e no aparelho de Golgi. A glicosilação pode ter um papel crítico na determinação da estrutura 3D dobrada final da proteína alvo. Em alguns casos, a glicosilação é necessária para o dobramento correto. A glicosilação N-ligada promove o dobramento de proteínas aumentando a solubilidade e medeia a ligação de proteínas a chaperonas de proteínas. As acompanhantes são proteínas responsáveis por dobrar e manter a estrutura de outras proteínas.
Existem basicamente dois tipos de glicosilação, glicosilação ligada a N e glicosilação ligada a O. A glicosilação ligada a N começa no retículo endoplasmático com a adição de um glicano precursor. O glicano precursor é modificado no aparelho de Golgi para produzir glicano complexo ligado covalentemente ao nitrogênio em um aminoácido asparagina. Em contraste, a glicosilação ligada a O é a adição covalente sequencial de açúcares individuais ao oxigênio nos aminoácidos serina e treonina dentro da estrutura da proteína madura.
Formação de ligações covalentes
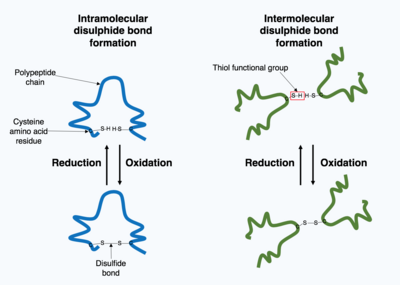
Muitas proteínas produzidas dentro da célula são secretadas fora da célula para funcionar como proteínas extracelulares. As proteínas extracelulares são expostas a uma ampla variedade de condições. Para estabilizar a estrutura da proteína 3D, as ligações covalentes são formadas dentro da proteína ou entre as diferentes cadeias polipeptídicas na estrutura quaternária. O tipo mais prevalente é uma ligação dissulfeto (também conhecida como ponte dissulfeto). Uma ligação dissulfeto é formada entre dois aminoácidos de cisteína usando seus grupos químicos de cadeia lateral contendo um átomo de enxofre, esses grupos químicos são conhecidos como grupos funcionais tiol. As pontes dissulfeto agem para estabilizar a estrutura pré-existente da proteína. As pontes dissulfeto são formadas em uma reação de oxidação entre dois grupos tiol e, portanto, precisam de um ambiente oxidante para reagir. Como resultado, as pontes dissulfeto são tipicamente formadas no ambiente oxidante do retículo endoplasmático, catalisadas por enzimas chamadas isomerases de dissulfeto de proteína. As pontes dissulfeto raramente são formadas no citoplasma, pois é um ambiente redutor.
Papel da síntese de proteínas na doença
Muitas doenças são causadas por mutações nos genes, devido à ligação direta entre a sequência de nucleotídeos do DNA e a sequência de aminoácidos da proteína codificada. Alterações na estrutura primária da proteína podem resultar em dobramento incorreto ou mau funcionamento da proteína. Mutações dentro de um único gene foram identificadas como causa de várias doenças, incluindo a doença falciforme, conhecida como distúrbios de gene único.
Doença falciforme
A doença falciforme é um grupo de doenças causadas por uma mutação em uma subunidade da hemoglobina, uma proteína encontrada nos glóbulos vermelhos responsável pelo transporte de oxigênio. A mais perigosa das doenças falciformes é conhecida como anemia falciforme. A anemia falciforme é o distúrbio de gene único recessivo homozigoto mais comum, o que significa que o indivíduo afetado deve carregar uma mutação em ambas as cópias do gene afetado (uma herdada de cada pai) para experimentar a doença. A hemoglobina tem uma estrutura quaternária complexa e é composta por quatro subunidades polipeptídicas - duas subunidades A e duas subunidades B. Pacientes com anemia falciforme têm uma mutação missense ou de substituição no gene que codifica a cadeia polipeptídica da subunidade B da hemoglobina. Uma mutação sem sentido significa que a mutação do nucleotídeo altera o trio de códons geral, de modo que um aminoácido diferente seja emparelhado com o novo códon. No caso da anemia falciforme, a mutação missense mais comum é uma mutação de nucleotídeo único de timina para adenina no gene da subunidade B da hemoglobina. Isso altera o códon 6 da codificação do aminoácido ácido glutâmico para a codificação da valina.
Essa mudança na estrutura primária da cadeia polipeptídica da subunidade B da hemoglobina altera a funcionalidade do complexo multi-subunidade da hemoglobina em condições de baixo oxigênio. Quando os glóbulos vermelhos descarregam oxigênio nos tecidos do corpo, a proteína da hemoglobina mutante começa a se unir para formar uma estrutura semi-sólida dentro do glóbulo vermelho. Isso distorce a forma do glóbulo vermelho, resultando na característica "foice" forma e reduz a flexibilidade celular. Este glóbulo vermelho rígido e distorcido pode se acumular nos vasos sanguíneos, criando um bloqueio. O bloqueio impede o fluxo sanguíneo para os tecidos e pode levar à morte do tecido, o que causa grande dor ao indivíduo.
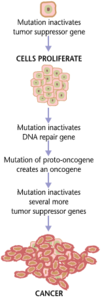
Câncer
Os cânceres se formam como resultado de mutações genéticas, bem como da tradução imprópria de proteínas. Além das células cancerígenas proliferarem anormalmente, elas suprimem a expressão de genes ou proteínas anti-apoptóticas ou pró-apoptóticas. A maioria das células cancerígenas vê uma mutação na proteína de sinalização Ras, que funciona como um transdutor de sinal on/off nas células. Nas células cancerígenas, a proteína RAS torna-se persistentemente ativa, promovendo assim a proliferação da célula devido à ausência de qualquer regulação. Além disso, a maioria das células cancerígenas carrega duas cópias mutantes do gene regulador p53, que atua como um porteiro para genes danificados e inicia a apoptose em células malignas. Na sua ausência, a célula não pode iniciar a apoptose ou sinalizar para que outras células a destruam.
À medida que as células tumorais proliferam, elas permanecem confinadas a uma área e são chamadas de benignas, ou tornam-se células malignas que migram para outras áreas do corpo. Muitas vezes, essas células malignas secretam proteases que quebram a matriz extracelular dos tecidos. Isso permite que o câncer entre em seu estágio terminal chamado Metástase, no qual as células entram na corrente sanguínea ou no sistema linfático para viajar para uma nova parte do corpo.