Bioinformática
Bioinformática () é um campo interdisciplinar que desenvolve métodos e ferramentas de software para entender dados biológicos, em particular quando os conjuntos de dados são grandes e complexos. Como um campo interdisciplinar da ciência, a bioinformática combina biologia, química, física, ciência da computação, engenharia da informação, matemática e estatística para analisar e interpretar os dados biológicos. A bioinformática tem sido usada para análises in silico de consultas biológicas usando técnicas computacionais e estatísticas.
Bioinformática inclui estudos biológicos que usam a programação de computadores como parte de sua metodologia, bem como "pipelines" que são usados repetidamente, particularmente no campo da genômica. Os usos comuns da bioinformática incluem a identificação de genes candidatos e polimorfismos de nucleotídeo único (SNPs). Freqüentemente, essa identificação é feita com o objetivo de entender melhor a base genética da doença, adaptações únicas, propriedades desejáveis (especialmente em espécies agrícolas) ou diferenças entre populações. De forma menos formal, a bioinformática também tenta entender os princípios organizacionais dentro das sequências de ácidos nucléicos e proteínas, chamados de proteômica.
O processamento de imagem e sinal permite a extração de resultados úteis de grandes quantidades de dados brutos. No campo da genética, auxilia no sequenciamento e anotação de genomas e suas mutações observadas. Ele desempenha um papel na mineração de texto da literatura biológica e no desenvolvimento de ontologias biológicas e genéticas para organizar e consultar dados biológicos. Também desempenha um papel na análise da expressão e regulação de genes e proteínas. As ferramentas de bioinformática auxiliam na comparação, análise e interpretação de dados genéticos e genômicos e, de forma mais geral, na compreensão dos aspectos evolutivos da biologia molecular. Em um nível mais integrador, ajuda a analisar e catalogar as vias e redes biológicas que são uma parte importante da biologia de sistemas. Na biologia estrutural, auxilia na simulação e modelagem de DNA, RNA, proteínas, bem como interações biomoleculares.
História
Historicamente, o termo bioinformática não significava o que significa hoje. Paulien Hogeweg e Ben Hesper o cunharam em 1970 para se referir ao estudo dos processos de informação em sistemas bióticos. Essa definição colocou a bioinformática como um campo paralelo à bioquímica (o estudo dos processos químicos em sistemas biológicos).
Sequências
Houve um tremendo avanço em velocidade e redução de custos desde a conclusão do Projeto Genoma Humano, com alguns laboratórios capazes de sequenciar mais de 100.000 bilhões de bases a cada ano, e um genoma completo pode ser sequenciado por mil dólares ou menos. Os computadores tornaram-se essenciais na biologia molecular quando as sequências de proteínas se tornaram disponíveis depois que Frederick Sanger determinou a sequência da insulina no início dos anos 1950. Comparar várias sequências manualmente acabou sendo impraticável. Uma pioneira no campo foi Margaret Oakley Dayhoff. Ela compilou um dos primeiros bancos de dados de sequências de proteínas, inicialmente publicado como livros e métodos pioneiros de alinhamento de sequências e evolução molecular. Outro colaborador inicial da bioinformática foi Elvin A. Kabat, que foi pioneiro na análise de sequências biológicas em 1970 com seus volumes abrangentes de sequências de anticorpos lançados com Tai Te Wu entre 1980 e 1991. Na década de 1970, novas técnicas de sequenciamento de DNA foram aplicadas ao bacteriófago MS2 e øX174, e as sequências de nucleotídeos estendidas foram então analisadas com algoritmos informativos e estatísticos. Esses estudos ilustraram que recursos bem conhecidos, como os segmentos de codificação e o código tripleto, são revelados em análises estatísticas diretas e, portanto, provam o conceito de que a bioinformática seria perspicaz.
Metas
Para estudar como as atividades celulares normais são alteradas em diferentes estados de doença, os dados biológicos devem ser combinados para formar uma imagem abrangente dessas atividades. Portanto, o campo da bioinformática evoluiu tanto que a tarefa mais premente agora envolve a análise e interpretação de vários tipos de dados. Isso também inclui sequências de nucleotídeos e aminoácidos, domínios de proteínas e estruturas de proteínas. O processo real de análise e interpretação de dados é referido como biologia computacional. Subdisciplinas importantes dentro da bioinformática e biologia computacional incluem:
- Desenvolvimento e implementação de programas de computador que permitem acesso eficiente, gestão e uso de, vários tipos de informação.
- Desenvolvimento de novos algoritmos (fórmulas matemáticos) e medidas estatísticas que avaliam as relações entre membros de grandes conjuntos de dados. Por exemplo, existem métodos para localizar um gene dentro de uma seqüência, prever a estrutura proteica e/ou função, e agrupar sequências proteicas em famílias de sequências relacionadas.
O principal objetivo da bioinformática é aumentar a compreensão dos processos biológicos. O que o diferencia de outras abordagens, no entanto, é seu foco no desenvolvimento e aplicação de técnicas computacionais intensivas para atingir esse objetivo. Os exemplos incluem: reconhecimento de padrões, mineração de dados, algoritmos de aprendizado de máquina e visualização. Os principais esforços de pesquisa no campo incluem alinhamento de sequências, descoberta de genes, montagem do genoma, design de drogas, descoberta de drogas, alinhamento da estrutura da proteína, previsão da estrutura da proteína, previsão da expressão gênica e interações proteína-proteína, estudos de associação do genoma, modelagem da evolução e divisão celular/mitose.
A bioinformática agora envolve a criação e o avanço de bancos de dados, algoritmos, técnicas computacionais e estatísticas e teoria para resolver problemas formais e práticos decorrentes do gerenciamento e análise de dados biológicos.
Nas últimas décadas, rápidos desenvolvimentos em genômica e outras tecnologias de pesquisa molecular e desenvolvimentos em tecnologias da informação se combinaram para produzir uma quantidade enorme de informações relacionadas à biologia molecular. Bioinformática é o nome dado a essas abordagens matemáticas e computacionais usadas para obter a compreensão dos processos biológicos.
Atividades comuns em bioinformática incluem mapeamento e análise de DNA e sequências de proteínas, alinhamento de DNA e sequências de proteínas para compará-las e criação e visualização de modelos 3-D de estruturas de proteínas.
Relação com outros campos
Bioinformática é um campo da ciência que é semelhante, mas distinto da computação biológica, embora muitas vezes seja considerado sinônimo de biologia computacional. A computação biológica usa a bioengenharia e a biologia para construir computadores biológicos, enquanto a bioinformática usa a computação para entender melhor a biologia. Bioinformática e biologia computacional envolvem a análise de dados biológicos, particularmente DNA, RNA e sequências de proteínas. O campo da bioinformática experimentou um crescimento explosivo a partir de meados da década de 1990, impulsionado em grande parte pelo Projeto Genoma Humano e pelos rápidos avanços na tecnologia de sequenciamento de DNA.
A análise de dados biológicos para produzir informações significativas envolve escrever e executar programas de software que usam algoritmos da teoria dos grafos, inteligência artificial, computação leve, mineração de dados, processamento de imagens e simulação de computador. Os algoritmos, por sua vez, dependem de fundamentos teóricos como matemática discreta, teoria de controle, teoria de sistemas, teoria da informação e estatística.
Análise de sequência
Desde que o fago Φ-X174 foi sequenciado em 1977, as sequências de DNA de milhares de organismos foram decodificadas e armazenadas em bancos de dados. Esta informação de sequência é analisada para determinar genes que codificam proteínas, genes de RNA, sequências regulatórias, motivos estruturais e sequências repetitivas. Uma comparação de genes dentro de uma espécie ou entre espécies diferentes pode mostrar semelhanças entre funções de proteínas ou relações entre espécies (o uso de sistemática molecular para construir árvores filogenéticas). Com a crescente quantidade de dados, há muito tempo tornou-se impraticável analisar manualmente as sequências de DNA. Programas de computador como o BLAST são usados rotineiramente para pesquisar sequências – desde 2008, de mais de 260.000 organismos, contendo mais de 190 bilhões de nucleotídeos.
Sequenciamento de DNA
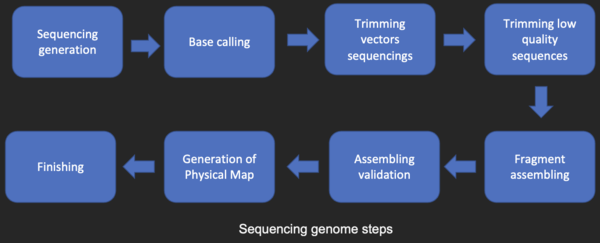
Antes que as sequências possam ser analisadas, elas devem ser obtidas do exemplo de banco de armazenamento de dados Genbank. O sequenciamento de DNA ainda é um problema não trivial, pois os dados brutos podem ser ruidosos ou afetados por sinais fracos. Algoritmos foram desenvolvidos para chamadas de base para as várias abordagens experimentais de sequenciamento de DNA.
Montagem de sequência
A maioria das técnicas de sequenciamento de DNA produz fragmentos curtos de sequência que precisam ser montados para obter sequências completas de genes ou genomas. A chamada técnica de sequenciamento shotgun (que foi usada, por exemplo, pelo The Institute for Genomic Research (TIGR) para sequenciar o primeiro genoma bacteriano, Haemophilus influenzae) gera as sequências de muitos milhares de pequenos DNA fragmentos (variando de 35 a 900 nucleotídeos de comprimento, dependendo da tecnologia de sequenciamento). As extremidades desses fragmentos se sobrepõem e, quando alinhadas adequadamente por um programa de montagem do genoma, podem ser usadas para reconstruir o genoma completo. O sequenciamento shotgun produz dados sequenciais rapidamente, mas a tarefa de montar os fragmentos pode ser bastante complicada para genomas maiores. Para um genoma tão grande quanto o genoma humano, pode levar muitos dias de tempo de CPU em computadores multiprocessadores de grande memória para montar os fragmentos, e a montagem resultante geralmente contém várias lacunas que devem ser preenchidas posteriormente. O sequenciamento shotgun é o método de escolha para virtualmente todos os genomas sequenciados (em vez de terminação de cadeia ou métodos de degradação química), e os algoritmos de montagem do genoma são uma área crítica da pesquisa em bioinformática.
Anotação do genoma
No contexto da genômica, a anotação é o processo de marcação dos genes e outras características biológicas em uma sequência de DNA. Esse processo precisa ser automatizado porque a maioria dos genomas é muito grande para anotar manualmente, sem falar no desejo de anotar o maior número possível de genomas, pois a taxa de sequenciamento deixou de ser um gargalo. A anotação é possível pelo fato de que os genes têm regiões de início e parada reconhecíveis, embora a sequência exata encontrada nessas regiões possa variar entre os genes.
A anotação do genoma pode ser classificada em três níveis: os níveis de nucleotídeo, proteína e processo.
A descoberta de genes é um aspecto principal da anotação em nível de nucleotídeo. Para genomas complexos, os métodos mais bem-sucedidos usam uma combinação de predição de genes ab initio e comparação de sequências com bancos de dados de sequências expressas e outros organismos. A anotação em nível de nucleotídeo também permite a integração da sequência do genoma com outros mapas genéticos e físicos do genoma.
O principal objetivo da anotação em nível de proteína é atribuir função aos produtos do genoma. Bancos de dados de sequências de proteínas e domínios e motivos funcionais são recursos poderosos para esse tipo de anotação. No entanto, metade das proteínas previstas em uma nova sequência do genoma tendem a não ter nenhuma função óbvia.
Entender a função dos genes e seus produtos no contexto da fisiologia celular e do organismo é o objetivo da anotação em nível de processo. Um dos obstáculos para esse nível de anotação tem sido a inconsistência de termos usados por diferentes sistemas de modelo. O Gene Ontology Consortium está ajudando a resolver esse problema.
A primeira descrição de um sistema abrangente de anotação de genoma foi publicada em 1995 pela equipe do The Institute for Genomic Research, que realizou o primeiro sequenciamento completo e análise do genoma de um organismo de vida livre, a bactéria Haemophilus influenzae . Owen White projetou e construiu um sistema de software para identificar os genes que codificam todas as proteínas, RNAs de transferência, RNAs ribossomais (e outros locais) e fazer atribuições funcionais iniciais. A maioria dos sistemas atuais de anotação de genoma funciona de forma semelhante, mas os programas disponíveis para análise de DNA genômico, como o programa GeneMark treinado e usado para encontrar genes codificadores de proteínas em Haemophilus influenzae, estão constantemente mudando e melhorando.
Seguindo os objetivos que o Projeto Genoma Humano deixou de alcançar após seu encerramento em 2003, surgiu um novo projeto desenvolvido pelo National Human Genome Research Institute nos EUA. O chamado projeto ENCODE é uma coleta colaborativa de dados dos elementos funcionais do genoma humano que usa tecnologias de sequenciamento de DNA de última geração e matrizes de ladrilhos genômicos, tecnologias capazes de gerar automaticamente grandes quantidades de dados a um custo por base drasticamente reduzido mas com a mesma precisão (erro de chamada de base) e fidelidade (erro de montagem).
Previsão da função genética
Embora a anotação do genoma seja baseada principalmente na similaridade da sequência (e, portanto, na homologia), outras propriedades das sequências podem ser usadas para prever a função dos genes. Na verdade, a maioria dos métodos de predição de funções de genes concentra-se em sequências de proteínas, pois são mais informativas e mais ricas em recursos. Por exemplo, a distribuição de aminoácidos hidrofóbicos prevê segmentos transmembrana em proteínas. No entanto, a previsão da função da proteína também pode usar informações externas, como dados de expressão de genes (ou proteínas), estrutura da proteína ou interações proteína-proteína.
Biologia evolutiva computacional
A biologia evolutiva é o estudo da origem e descendência das espécies, bem como sua mudança ao longo do tempo. A informática ajudou os biólogos evolutivos, permitindo que os pesquisadores:
- traçar a evolução de um grande número de organismos medindo mudanças em seu DNA, em vez de através da taxonomia física ou apenas observações fisiológicas,
- comparar genomas inteiros, o que permite o estudo de eventos evolutivos mais complexos, como duplicação de genes, transferência de genes horizontais e a previsão de fatores importantes na especiação bacteriana,
- construir modelos complexos de genética de população computacional para prever o resultado do sistema ao longo do tempo
- rastrear e compartilhar informações sobre um número cada vez maior de espécies e organismos
Trabalhos futuros se esforçam para reconstruir a agora mais complexa árvore da vida.
A área de pesquisa dentro da ciência da computação que usa algoritmos genéticos às vezes é confundida com a biologia evolutiva computacional, mas as duas áreas não estão necessariamente relacionadas.
Genômica comparativa
O núcleo da análise comparativa do genoma é o estabelecimento da correspondência entre genes (análise de ortologia) ou outras características genômicas em diferentes organismos. São esses mapas intergenómicos que permitem traçar os processos evolutivos responsáveis pela divergência de dois genomas. Uma infinidade de eventos evolutivos atuando em vários níveis organizacionais moldam a evolução do genoma. No nível mais baixo, as mutações pontuais afetam nucleotídeos individuais. Em um nível superior, grandes segmentos cromossômicos sofrem duplicação, transferência lateral, inversão, transposição, deleção e inserção. Em última análise, genomas inteiros estão envolvidos em processos de hibridação, poliploidização e endossimbiose, muitas vezes levando a uma rápida especiação. A complexidade da evolução do genoma coloca muitos desafios emocionantes para os desenvolvedores de modelos e algoritmos matemáticos, que recorrem a um espectro de técnicas algorítmicas, estatísticas e matemáticas, variando de algoritmos exatos, heurísticos, de parâmetros fixos e de aproximação para problemas baseados em modelos de parcimônia até Markov cadeia de algoritmos de Monte Carlo para análise Bayesiana de problemas baseados em modelos probabilísticos.
Muitos desses estudos são baseados na detecção de homologia de sequência para atribuir sequências a famílias de proteínas.
Pangenômica
Pangenômica é um conceito introduzido em 2005 por Tettelin e Medini, que eventualmente se enraizou na bioinformática. O genoma pan é o repertório gênico completo de um grupo taxonômico particular: embora inicialmente aplicado a cepas estreitamente relacionadas de uma espécie, pode ser aplicado a um contexto maior como gênero, filo, etc. É dividido em duas partes- O genoma central: Conjunto de genes comuns a todos os genomas em estudo (geralmente são genes de manutenção vitais para a sobrevivência) e O Genoma Dispensável/Flexível: Conjunto de genes não presentes em todos, exceto em um ou alguns genomas em estudo. Uma ferramenta de bioinformática BPGA pode ser usada para caracterizar o Pan Genome de espécies bacterianas.
Genética da doença
Com o advento do sequenciamento de próxima geração, estamos obtendo dados de sequência suficientes para mapear os genes de doenças complexas, incluindo infertilidade, câncer de mama ou doença de Alzheimer. Os estudos de associação do genoma são uma abordagem útil para identificar as mutações responsáveis por tais doenças complexas. Por meio desses estudos, foram identificadas milhares de variantes de DNA associadas a doenças e características semelhantes. Além disso, a possibilidade de os genes serem usados no prognóstico, diagnóstico ou tratamento é uma das aplicações mais essenciais. Muitos estudos estão discutindo tanto as maneiras promissoras de escolher os genes a serem usados quanto os problemas e armadilhas do uso de genes para prever a presença de doenças ou o prognóstico.
Estudos de associação em todo o genoma identificaram com sucesso milhares de variantes genéticas comuns para doenças e características complexas; no entanto, essas variantes comuns explicam apenas uma pequena fração da herdabilidade. Variantes raras podem ser responsáveis por parte da hereditariedade ausente. Estudos de sequenciamento de genoma inteiro em grande escala sequenciaram rapidamente milhões de genomas inteiros, e esses estudos identificaram centenas de milhões de variantes raras. As anotações funcionais preveem o efeito ou a função de uma variante genética e ajudam a priorizar variantes funcionais raras, e a incorporação dessas anotações pode efetivamente aumentar o poder da associação genética da análise de variantes raras de estudos de sequenciamento do genoma inteiro. Algumas ferramentas foram desenvolvidas para fornecer análise de associação de variante rara completa para dados de sequenciamento de genoma completo, incluindo integração de dados de genótipo e suas anotações funcionais, análise de associação, resumo de resultados e visualização. A meta-análise de estudos de sequenciamento do genoma inteiro fornece uma solução atraente para o problema de coletar grandes tamanhos de amostra para descobrir variantes raras associadas a fenótipos complexos.
Análise de mutações no câncer
No câncer, os genomas das células afetadas são rearranjados de forma complexa ou mesmo imprevisível. Esforços maciços de sequenciamento são usados para identificar mutações pontuais previamente desconhecidas em uma variedade de genes no câncer. Os bioinformáticos continuam a produzir sistemas automatizados especializados para gerenciar o grande volume de dados de sequência produzidos e criam novos algoritmos e software para comparar os resultados do sequenciamento com a crescente coleção de sequências do genoma humano e polimorfismos da linhagem germinativa. Novas tecnologias de detecção física são empregadas, como microarranjos de oligonucleotídeos para identificar ganhos e perdas cromossômicas (denominado hibridação genômica comparativa) e arranjos de polimorfismo de nucleotídeo único para detectar mutações pontuais conhecidas. Esses métodos de detecção medem simultaneamente várias centenas de milhares de locais em todo o genoma e, quando usados em alto rendimento para medir milhares de amostras, geram terabytes de dados por experimento. Mais uma vez, as enormes quantidades e novos tipos de dados geram novas oportunidades para os bioinformáticos. Freqüentemente, os dados contêm variabilidade considerável ou ruído e, portanto, o modelo oculto de Markov e os métodos de análise de ponto de mudança estão sendo desenvolvidos para inferir mudanças reais no número de cópias.
Dois princípios importantes podem ser usados na análise bioinformática de genomas de câncer relacionados à identificação de mutações no exoma. Primeiro, o câncer é uma doença de mutações somáticas acumuladas nos genes. O segundo câncer contém mutações de motorista que precisam ser diferenciadas de passageiros.
Com os avanços que esta tecnologia de sequenciamento de última geração está proporcionando ao campo da bioinformática, a genômica do câncer pode mudar drasticamente. Esses novos métodos e software permitem que os bioinformáticos sequenciem muitos genomas de câncer de forma rápida e acessível. Isso poderia criar um processo mais flexível para classificar os tipos de câncer por meio da análise de mutações causadas pelo câncer no genoma. Além disso, o rastreamento de pacientes enquanto a doença progride pode ser possível no futuro com a sequência de amostras de câncer.
Outro tipo de dados que requer um novo desenvolvimento de informática é a análise de lesões recorrentes entre muitos tumores.
Expressão de genes e proteínas
Análise da expressão gênica
A expressão de muitos genes pode ser determinada medindo os níveis de mRNA com várias técnicas, incluindo microarrays, sequenciamento de marca de sequência de cDNA expresso (EST), análise serial de sequenciamento de marca de expressão gênica (SAGE), sequenciamento de assinatura massivamente paralelo (MPSS), RNA -Seq, também conhecido como "Sequenciamento completo de espingarda de transcriptoma" (WTSS), ou várias aplicações de hibridização in-situ multiplexada. Todas essas técnicas são extremamente propensas a ruído e/ou sujeitas a viés na medição biológica, e uma importante área de pesquisa em biologia computacional envolve o desenvolvimento de ferramentas estatísticas para separar o sinal do ruído em estudos de expressão gênica de alto rendimento. Tais estudos são frequentemente usados para determinar os genes implicados em um distúrbio: pode-se comparar dados de microarray de células epiteliais cancerígenas com dados de células não cancerígenas para determinar os transcritos que são regulados para cima e para baixo em uma determinada população de células cancerígenas..
Análise da expressão de proteínas
Microarrays de proteínas e espectrometria de massa (MS) de alto rendimento (HT) podem fornecer um instantâneo das proteínas presentes em uma amostra biológica. A bioinformática está muito envolvida em dar sentido ao microarranjo de proteínas e aos dados do HT MS; a primeira abordagem enfrenta problemas semelhantes aos dos microarrays direcionados ao mRNA, a última envolve o problema de combinar grandes quantidades de dados de massa com massas previstas de bancos de dados de sequências de proteínas e a complicada análise estatística de amostras em que vários, mas peptídeos incompletos de cada proteína são detectou. A localização de proteínas celulares em um contexto de tecido pode ser obtida por meio de proteômica de afinidade exibida como dados espaciais com base em imuno-histoquímica e microarranjos de tecidos.
Análise da regulamentação
A regulação gênica é a orquestração complexa de eventos pelos quais um sinal, potencialmente um sinal extracelular como um hormônio, eventualmente leva a um aumento ou diminuição da atividade de uma ou mais proteínas. Técnicas de bioinformática têm sido aplicadas para explorar várias etapas desse processo.
Por exemplo, a expressão gênica pode ser regulada por elementos próximos no genoma. A análise do promotor envolve a identificação e o estudo de motivos de sequência no DNA ao redor da região codificadora de um gene. Esses motivos influenciam a extensão em que essa região é transcrita em mRNA. Elementos intensificadores distantes do promotor também podem regular a expressão gênica, por meio de interações tridimensionais em loop. Essas interações podem ser determinadas por análise bioinformática de experimentos de captura de conformação cromossômica.
Dados de expressão podem ser usados para inferir a regulação de genes: pode-se comparar dados de microarray de uma ampla variedade de estados de um organismo para formar hipóteses sobre os genes envolvidos em cada estado. Em um organismo unicelular, pode-se comparar os estágios do ciclo celular, juntamente com várias condições de estresse (choque térmico, fome, etc.). Pode-se então aplicar algoritmos de agrupamento a esses dados de expressão para determinar quais genes são co-expressos. Por exemplo, as regiões upstream (promotoras) de genes co-expressos podem ser pesquisadas em busca de elementos reguladores super-representados. Exemplos de algoritmos de agrupamento aplicados no agrupamento de genes são agrupamento k-means, mapas auto-organizados (SOMs), agrupamento hierárquico e métodos de agrupamento de consenso.
Análise da organização celular
Várias abordagens foram desenvolvidas para analisar a localização de organelas, genes, proteínas e outros componentes dentro das células. Isso é relevante, pois a localização desses componentes afeta os eventos dentro de uma célula e, portanto, nos ajuda a prever o comportamento dos sistemas biológicos. Uma categoria de ontologia genética, componente celular, foi criada para capturar a localização subcelular em muitos bancos de dados biológicos.
Microscopia e análise de imagem
Imagens microscópicas nos permitem localizar tanto organelas quanto moléculas. Também pode nos ajudar a distinguir entre células normais e anormais, por ex. em câncer.
Localização de proteínas
A localização das proteínas nos ajuda a avaliar o papel de uma proteína. Por exemplo, se uma proteína for encontrada no núcleo, ela pode estar envolvida na regulação ou splicing de genes. Por outro lado, se uma proteína for encontrada nas mitocôndrias, ela pode estar envolvida na respiração ou em outros processos metabólicos. A localização da proteína é, portanto, um componente importante da previsão da função da proteína. Existem recursos de previsão de localização subcelular de proteínas bem desenvolvidos disponíveis, incluindo bancos de dados de localização subcelular de proteínas e ferramentas de previsão.
Organização nuclear da cromatina
Dados de experimentos de captura de conformação cromossômica de alto rendimento, como Hi-C (experimento) e ChIA-PET, podem fornecer informações sobre a proximidade espacial de loci de DNA. A análise desses experimentos pode determinar a estrutura tridimensional e a organização nuclear da cromatina. Os desafios da bioinformática neste campo incluem o particionamento do genoma em domínios, como Domínios de Associação Topológica (TADs), que são organizados juntos no espaço tridimensional.
Bioinformática estrutural
A previsão da estrutura da proteína é outra aplicação importante da bioinformática. A sequência de aminoácidos de uma proteína, a chamada estrutura primária, pode ser facilmente determinada a partir da sequência do gene que a codifica. Na grande maioria dos casos, essa estrutura primária determina exclusivamente uma estrutura em seu ambiente nativo. (Existem exceções, como o príon da encefalopatia espongiforme bovina (doença da vaca louca).) O conhecimento dessa estrutura é vital para entender a função da proteína. A informação estrutural é geralmente classificada como estrutura secundária, terciária e quaternária. Uma solução geral viável para tais previsões permanece um problema em aberto. A maioria dos esforços até agora foram direcionados para heurísticas que funcionam na maior parte do tempo.
Uma das ideias-chave em bioinformática é a noção de homologia. No ramo genômico da bioinformática, a homologia é usada para prever a função de um gene: se a sequência do gene A, cuja função é conhecida, é homóloga à sequência do gene B, cuja função é desconhecida, pode-se inferir que B pode compartilhar a função de A. No ramo estrutural da bioinformática, a homologia é usada para determinar quais partes de uma proteína são importantes na formação da estrutura e na interação com outras proteínas. Em uma técnica chamada modelagem de homologia, essa informação é usada para prever a estrutura de uma proteína uma vez que a estrutura de uma proteína homóloga é conhecida. Até recentemente, esta permaneceu a única maneira de prever estruturas de proteínas de forma confiável. No entanto, um avanço revolucionário ocorreu com o lançamento de um novo software baseado em algoritmos de aprendizado profundo chamado AlphaFold, desenvolvido por uma equipe de bioinformática dentro da IA do Google. departamento de pesquisa DeepMind. O AlphaFold, durante a 14ª Avaliação Crítica da Previsão de Estrutura de Proteínas (CASP14), competição computacional de software de previsão de estruturas de proteínas, tornou-se o primeiro candidato a fornecer envios de predições com precisão competitiva com estruturas experimentais na maioria dos casos e superando em muito todos os outros métodos de software de predição até a esse ponto. Desde então, o AlphaFold liberou as estruturas previstas para centenas de milhões de proteínas.
Um exemplo disso é a hemoglobina em humanos e a hemoglobina em leguminosas (leg-hemoglobina), que são parentes distantes da mesma superfamília de proteínas. Ambos têm a mesma finalidade de transportar oxigênio no organismo. Embora ambas as proteínas tenham sequências de aminoácidos completamente diferentes, suas estruturas protéicas são virtualmente idênticas, o que reflete seus propósitos quase idênticos e ancestral compartilhado.
Outras técnicas para prever a estrutura da proteína incluem o encadeamento de proteínas e a modelagem baseada em física de novo (do zero).
Outro aspecto da bioinformática estrutural inclui o uso de estruturas de proteínas para modelos de triagem virtual, como modelos quantitativos de relação estrutura-atividade e modelos proteoquimométricos (PCM). Além disso, a estrutura cristalina de uma proteína pode ser usada na simulação de, por exemplo, estudos de ligação de ligantes e estudos de mutagênese in silico.
Biologia de redes e sistemas
Análise de rede procura entender as relações dentro de redes biológicas, como redes metabólicas ou de interação proteína-proteína. Embora as redes biológicas possam ser construídas a partir de um único tipo de molécula ou entidade (como genes), a biologia de rede geralmente tenta integrar muitos tipos de dados diferentes, como proteínas, pequenas moléculas, dados de expressão gênica e outros, todos conectados fisicamente, funcionalmente ou ambos.
Biologia de sistemas envolve o uso de simulações de computador de subsistemas celulares (como as redes de metabólitos e enzimas que compreendem o metabolismo, vias de transdução de sinal e redes reguladoras de genes) para analisar e visualizar as conexões complexas desses processos celulares. A vida artificial ou evolução virtual tenta entender os processos evolutivos por meio da simulação por computador de formas de vida simples (artificiais).
Redes de interação molecular
Dezenas de milhares de estruturas tridimensionais de proteínas foram determinadas por cristalografia de raios-X e espectroscopia de ressonância magnética nuclear de proteínas (RMN de proteínas) e uma questão central em bioinformática estrutural é se é prático prever apenas possíveis interações proteína-proteína com base nessas formas 3D, sem realizar experimentos de interação proteína-proteína. Uma variedade de métodos foi desenvolvida para resolver o problema de docking proteína-proteína, embora pareça que ainda há muito trabalho a ser feito neste campo.
Outras interações encontradas no campo incluem proteína-ligante (incluindo droga) e proteína-peptídeo. A simulação dinâmica molecular do movimento de átomos sobre ligações rotativas é o princípio fundamental por trás dos algoritmos computacionais, denominados algoritmos de encaixe, para estudar as interações moleculares.
Outros
Análise da literatura
O crescimento do número de literatura publicada torna virtualmente impossível ler todos os artigos, resultando em subcampos de pesquisa desconexos. A análise da literatura visa empregar linguística computacional e estatística para explorar essa crescente biblioteca de recursos de texto. Por exemplo:
- Reconhecimento de abreviação – identificar a longa forma e abreviação de termos biológicos
- Reconhecimento de denominação – reconhecendo termos biológicos como nomes genéticos
- Interação da proteína – identificar quais proteínas interagem com as quais as proteínas do texto
A área de pesquisa baseia-se na estatística e na linguística computacional.
Análise de imagem de alto rendimento
As tecnologias computacionais são usadas para acelerar ou automatizar totalmente o processamento, quantificação e análise de grandes quantidades de imagens biomédicas com alto conteúdo de informação. Os sistemas modernos de análise de imagens aumentam a capacidade de um observador de fazer medições a partir de um conjunto grande ou complexo de imagens, melhorando a precisão, a objetividade ou a velocidade. Um sistema de análise totalmente desenvolvido pode substituir completamente o observador. Embora esses sistemas não sejam exclusivos da imagem biomédica, a imagem biomédica está se tornando mais importante tanto para diagnóstico quanto para pesquisa. Alguns exemplos são:
- quantificação de alta produtividade e alta fidelidade e localização subcelular (elevação de alto conteúdo, citoistopatologia, informática de bioimagem)
- morfometria
- análise de imagem clínica e visualização
- determinar os padrões de fluxo de ar em tempo real em pulmões respiratórios de animais vivos
- quantificando o tamanho da oclusão em imagens em tempo real do desenvolvimento e recuperação durante a lesão arterial
- fazendo observações comportamentais de gravações de vídeo estendidas de animais de laboratório
- medidas infravermelhas para determinação da atividade metabólica
- inferindo sobreposições de clones no mapeamento de DNA, por exemplo, a pontuação de Sulston
Análise de dados de célula única de alto rendimento
Técnicas computacionais são usadas para analisar dados de células únicas de alto rendimento e baixa medição, como os obtidos por citometria de fluxo. Esses métodos normalmente envolvem encontrar populações de células que são relevantes para um determinado estado de doença ou condição experimental.
Informática da biodiversidade
A informática da biodiversidade lida com a coleta e análise de dados de biodiversidade, como bancos de dados taxonômicos ou dados de microbioma. Exemplos de tais análises incluem filogenética, modelagem de nicho, mapeamento de riqueza de espécies, código de barras de DNA ou ferramentas de identificação de espécies.
Ontologias e integração de dados
Ontologias biológicas são grafos acíclicos direcionados de vocabulários controlados. Eles são projetados para capturar conceitos e descrições biológicas de forma que possam ser facilmente categorizados e analisados com computadores. Quando categorizados dessa forma, é possível obter valor agregado da análise holística e integrada.
O OBO Foundry foi um esforço para padronizar certas ontologias. Uma das mais difundidas é a ontologia Gene que descreve a função do gene. Existem também ontologias que descrevem fenótipos.
Bancos de dados
Bancos de dados são essenciais para pesquisas e aplicações em bioinformática. Existem muitos bancos de dados, cobrindo vários tipos de informações: por exemplo, sequências de DNA e proteínas, estruturas moleculares, fenótipos e biodiversidade. Os bancos de dados podem conter dados empíricos (obtidos diretamente de experimentos), dados previstos (obtidos por análise) ou, mais comumente, ambos. Eles podem ser específicos para um determinado organismo, via ou molécula de interesse. Como alternativa, eles podem incorporar dados compilados de vários outros bancos de dados. Esses bancos de dados variam em seu formato, mecanismo de acesso e se são públicos ou não.
Alguns dos bancos de dados mais usados estão listados abaixo. Para uma lista mais abrangente, consulte o link no início da subseção.
- Usado em análise de sequência biológica: Genbank, UniProt
- Usado na análise da estrutura: Banco de Dados de Proteína (PDB)
- Usado para encontrar Famílias de Proteína e Motivo Encontrar: InterPro, Pfam
- Usado para a próxima geração Sequencing: Sequence Read Archive
- Usado em Análise de Rede: Bases de dados Metabolic Pathway (KEGG, BioCyc), bancos de dados de análise de interação, redes funcionais
- Usado na concepção de circuitos genéticos sintéticos: GenoCAD
Software e ferramentas
As ferramentas de software para bioinformática variam de simples ferramentas de linha de comando a programas gráficos mais complexos e serviços da Web independentes disponíveis em várias empresas de bioinformática ou instituições públicas.
Software de bioinformática de código aberto
Muitas ferramentas de software livre e de código aberto existiram e continuaram a crescer desde a década de 1980. A combinação de uma necessidade contínua de novos algoritmos para a análise de tipos emergentes de leituras biológicas, o potencial para experimentos in silico inovadores e bases de código aberto disponíveis gratuitamente ajudaram a criar oportunidades para todos os grupos de pesquisa contribuir tanto para a bioinformática quanto para a gama de software de código aberto disponível, independentemente de seus acordos de financiamento. As ferramentas de código aberto geralmente atuam como incubadoras de ideias ou plug-ins suportados pela comunidade em aplicativos comerciais. Eles também podem fornecer padrões de fato e modelos de objetos compartilhados para auxiliar no desafio da integração da bioinformação.
A gama de pacotes de software de código aberto inclui títulos como Bioconductor, BioPerl, Biopython, BioJava, BioJS, BioRuby, Bioclipse, EMBOSS,.NET Bio, Orange com seu complemento de bioinformática, Apache Taverna, UGENE e GenoCAD. Para manter essa tradição e criar mais oportunidades, a Open Bioinformatics Foundation, sem fins lucrativos, apoia a Conferência Anual de Código Aberto de Bioinformática (BOSC) desde 2000.
Serviços Web em bioinformática
As interfaces baseadas em SOAP e REST foram desenvolvidas para uma ampla variedade de aplicativos de bioinformática, permitindo que um aplicativo executado em um computador em uma parte do mundo use algoritmos, dados e recursos de computação em servidores em outras partes do mundo. As principais vantagens derivam do fato de que os usuários finais não precisam lidar com despesas gerais de manutenção de software e banco de dados.
Os serviços básicos de bioinformática são classificados pela EBI em três categorias: SSS (Sequence Search Services), MSA (Multiple Sequence Alignment) e BSA (Biological Sequence Analysis). A disponibilidade desses recursos de bioinformática orientados a serviços demonstra a aplicabilidade de soluções de bioinformática baseadas na web e vão desde uma coleção de ferramentas autônomas com um formato de dados comum em uma interface única, autônoma ou baseada na web, até bioinformática integrativa, distribuída e extensível sistemas de gerenciamento de fluxo de trabalho.
Sistemas de gerenciamento de fluxo de trabalho de bioinformática
Um sistema de gerenciamento de fluxo de trabalho de bioinformática é uma forma especializada de um sistema de gerenciamento de fluxo de trabalho projetado especificamente para compor e executar uma série de etapas computacionais ou de manipulação de dados, ou um fluxo de trabalho, em um aplicativo de bioinformática. Tais sistemas são projetados para
- fornecer um ambiente fácil de usar para os próprios cientistas de aplicativos individuais criarem seus próprios fluxos de trabalho,
- fornecer ferramentas interativas para os cientistas que lhes permitem executar seus fluxos de trabalho e visualizar seus resultados em tempo real,
- simplificar o processo de compartilhamento e reutilização de fluxos de trabalho entre os cientistas e
- habilitar os cientistas a rastrear a comprovação dos resultados de execução de fluxo de trabalho e as etapas de criação de fluxo de trabalho.
Algumas das plataformas que oferecem este serviço: Galaxy, Kepler, Taverna, UGENE, Anduril, HIVE.
BioCompute e Objetos de BioCompute
Em 2014, a Food and Drug Administration dos EUA patrocinou uma conferência realizada no National Institutes of Health Bethesda Campus para discutir a reprodutibilidade em bioinformática. Nos três anos seguintes, um consórcio de partes interessadas se reuniu regularmente para discutir o que se tornaria o paradigma da BioCompute. Essas partes interessadas incluíam representantes do governo, da indústria e de entidades acadêmicas. Os líderes da sessão representaram várias filiais dos institutos e centros da FDA e do NIH, entidades sem fins lucrativos, incluindo o Human Variome Project e a European Federation for Medical Informatics, e instituições de pesquisa, incluindo Stanford, o New York Genome Center e a George Washington University.
Decidiu-se que o paradigma BioCompute seria na forma de 'cadernos de laboratório' que permitem a reprodutibilidade, replicação, revisão e reutilização de protocolos de bioinformática. Isso foi proposto para permitir maior continuidade dentro de um grupo de pesquisa ao longo do fluxo normal de pessoal, ao mesmo tempo em que promove a troca de ideias entre os grupos. O FDA dos EUA financiou esse trabalho para que as informações sobre os oleodutos fossem mais transparentes e acessíveis para sua equipe reguladora.
Em 2016, o grupo se reuniu novamente no NIH em Bethesda e discutiu o potencial de um Objeto BioCompute, uma instância do paradigma BioCompute. Este trabalho foi copiado como um "uso de avaliação padrão" documento e um documento pré-impresso carregado no bioRxiv. O objeto BioCompute permite que o registro JSON seja compartilhado entre funcionários, colaboradores e reguladores.
Plataformas de educação
Assim como os cursos presenciais de mestrado ministrados em muitas universidades, a natureza computacional da bioinformática a empresta para o aprendizado on-line e assistido por computador. Plataformas de software projetadas para ensinar conceitos e métodos de bioinformática incluem Rosalind e cursos online oferecidos pelo Portal de Treinamento do Instituto Suíço de Bioinformática. Os Canadian Bioinformatics Workshops fornecem vídeos e slides de workshops de treinamento em seu site sob uma licença Creative Commons. O projeto 4273π ou projeto 4273pi também oferece materiais educacionais de código aberto gratuitamente. O curso é executado em computadores Raspberry Pi de baixo custo e tem sido usado para ensinar adultos e alunos de escolas. O 4273π é desenvolvido ativamente por um consórcio de acadêmicos e equipes de pesquisa que executam bioinformática em nível de pesquisa usando computadores Raspberry Pi e o sistema operacional 4273π.
As plataformas MOOC também fornecem certificações on-line em bioinformática e disciplinas relacionadas, incluindo a Especialização em Bioinformática do Coursera (UC San Diego) e a Especialização em Ciência de Dados Genômicos (Johns Hopkins), bem como a Análise de Dados da EdX para Ciências da Vida Série X (Harvard).
Conferências
Existem várias grandes conferências que tratam da bioinformática. Alguns dos exemplos mais notáveis são Sistemas Inteligentes para Biologia Molecular (ISMB), Conferência Europeia sobre Biologia Computacional (ECCB) e Pesquisa em Biologia Molecular Computacional (RECOMB).
Contenido relacionado
Kremvax
Apicomplexa
Modelo de Objeto de Documento